Réparation de l’ADN
Introduction :
Les dommages à l’ADN intervenant spontanément et résultant d’agents de l’environnement existant en permanence, la plupart des organismes possèdent des capacités à réparer leur ADN.
L’ADN est la seule macromolécule à être réparée par les cellules.
Les mécanismes sont de trois types :
La restauration de la zone endommagée : c’est le plus simple.
Une action enzymatique restaure la structure sans casser le squelette
La suppression de la zone endommagée : les bases ou le groupe de nucléotides incorrects sont remplacés.
La tolérance de la zone endommagée : il n’y a pas de réparation ; le dommage est accepté.
La restauration de la zone endommagée :
La photo-réactivation : (plantes, bactéries et champignon) :
Il est la fois le plus simple et sans doute le plus ancien des systèmes de réparation.
Une simple enzyme la photolyase peut dissocier les dimères de thymine induite par les UV solaires (en cassant les liaisons covalentes) en présence de lumière. (Fig.1).

Figure 1 : La photo –réaction
La réparation des ruptures sur un seul brin :
Les rayons X et quelques produits chimiques comme les peroxydes peuvent causer des ruptures dans le squelette de l’ADN.
Les ruptures sur un seul brin sont rapidement réparées par une ligase. (Fig.2).
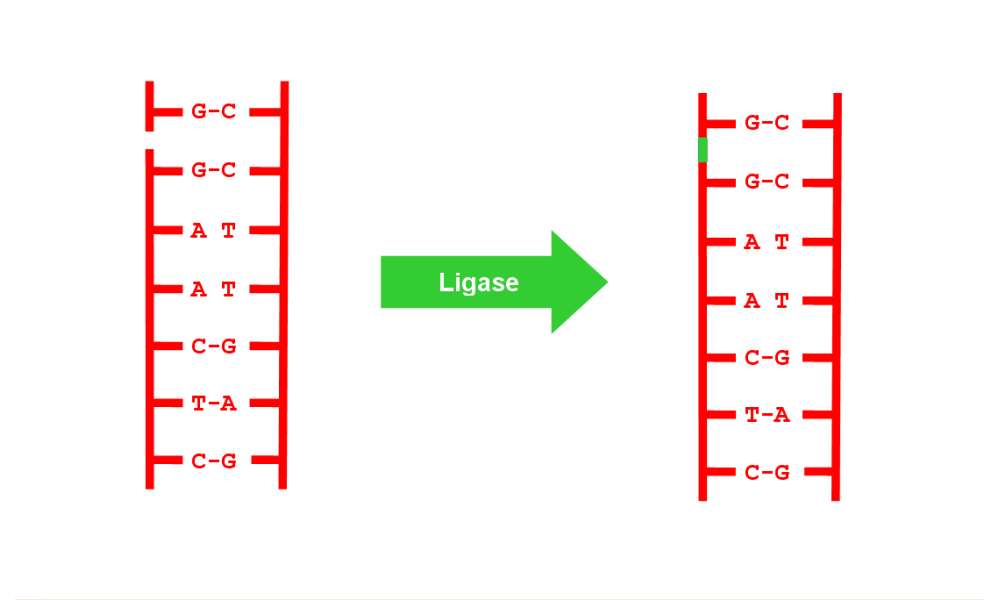
Figure 2 : réparation de la rupture sur un seul brin
La suppression de la zone endommagée :
La réparation par excision de bases : (Système BER) : (Fig. 3) :
Ce mécanisme est présent chez les procaryotes et les eucaryotes et essentiellement impliqué dans les réparations de mutations endogènes jusqu’à 4 nucléotides.
Le système BER permet l’élimination des bases anormales
La base inappropriée est détachée de sa liaison avec le sucre et remplacée.
Ce sont des glycosylases qui rompent la liaison.
Par exemple l’uracile-glycosylase sépare l’uracile de l’ADN.
L’uracile n’est pas habituellement dans l’ADN, mais peut exister si la cytosine est déaminée
L’enzyme reconnaît l’uracile et coupe la liaison N-glycosidique avec le déoxyribose.
La base est alors excisée et une nouvelle base est insérée par la polymérase en utilisant le deuxième brin comme matrice.
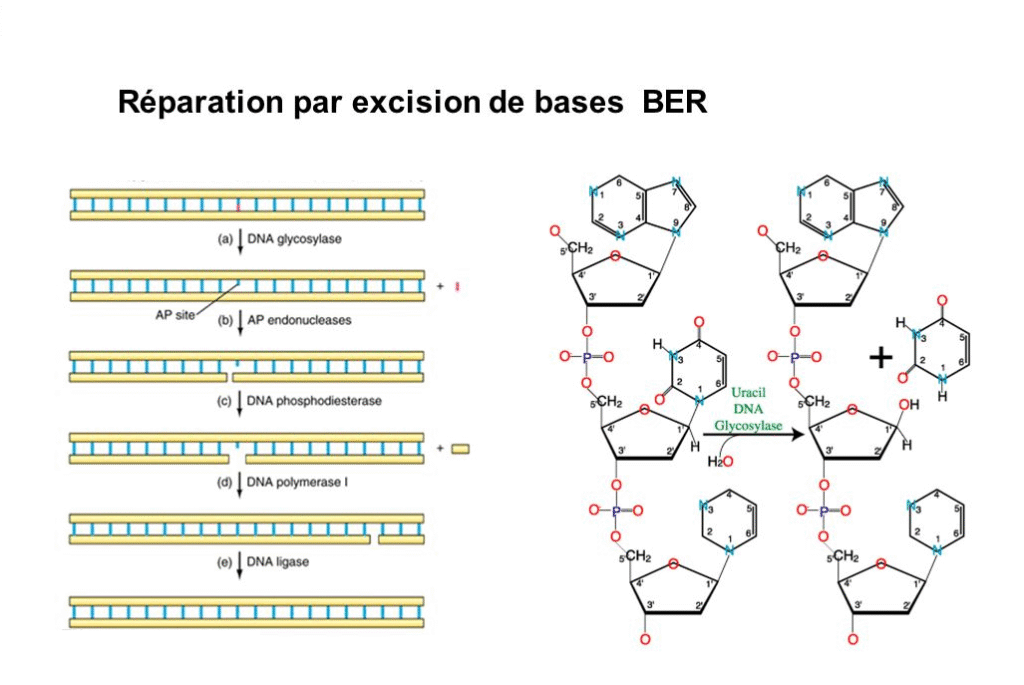
Figure 3 :
Réparation de mésappariements par le système Mut HLS :
Ce mécanisme est également présent chez les procaryotes et les eucaryotes et est post-réplicatif.
Il permet la réparation des erreurs d’appariement entre les chaînes d’ADN après la réplication.
Il est réalisé par un groupe de protéines qui peuvent “scanner” l’ADN et détecter les paires de bases incorrectes ou mal appariées.
Le nucléotide incorrect est supprimé et l’ADN polymérase opère un deuxième passage pour rétablir la séquence convenable.
Des protéines réparatrices de mauvais appariements ont été récemment identifiées chez l’Homme.
Elles sont très semblables à celles de l’Escherichia Coli et la levure. (Fig.4)

Figure 4 : Réparation du mésappariement par le système Mut HLS
La réparation par excision de nucléotides : (Système NER) :
Ce mécanisme intervient sur les dommages importants de l’ADN qui créent des blocages de la réplication et de la transcription.
Elle se déroule en 3 étapes successives :
Un complexe enzymatique qui comporte une exonucléase enlève un oligo nucléotide du brin à réparer.
Ce complexe nécessite la présence des protéines spécifiques dont l’une d’elles sont absentes ou modifiées dans le Xéroderma pigmentosum.
Les nucléotides manquants sont remplacés par l’ADN polymérase qui utilise le brin complémentaire comme modèle.
Une ligase assure la continuité. (Fig.5).

Figure 5 :
La tolérance de la zone endommagée :
Tous les dommages de l’ADN ne peuvent être réparés immédiatement.
Certains persistent.
Si un œil de réplication contient des altérations (par exemple un dimère de pyrimidine), il y a normalement blocage de la réplication.
Cependant, chez les Eucaryotes, la réplication peut être initiée à de nombreux endroits.
Elle peut donc reprendre au delà du dimère, laissant un secteur de la molécule non répliqué.
Ceci peut avoir des conséquences importantes en cas de division cellulaire.
Toutefois, il y a un moyen de réparation par recombinaison avec l’autre chromatide ou la chromatide sœur, ce qui aboutit à deux molécules sœur intactes, l’une d’elle contenant encore le dimère. (Fig.6).
La réparation par recombinaison entre molécules-filles:
La réparation par recombinaison correspond à la synthèse trans-lésionnelle (TLS) qui consiste à poursuivre la réplication de l’ADN au niveau d’une lésion du brin matriciel de l’ADN ne permettant aucun appariement.
Elle se réalise en même temps que la réplication.
L’ADN-polymérase II a un rôle important dans la reprise de la synthèse d’ADN après la lésion, l’ADN-polymérase III est alors transitoirement expulsée pour que la réplication puisse continuer.
L’ADN-polymérase III arrive au niveau d’une erreur et est éjectée par les enzymes de la TLS qui remplacent la base erronée ou alors qui permet la poursuite de la réplication un peu plus loin (ceci suivant l’ADN-polymérase utilisé (II, IV ou V)).

Figure 6 :
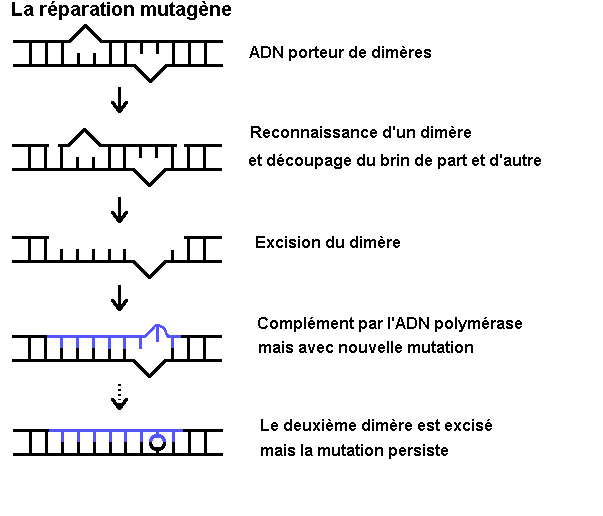
La réparation mutagène :
Un scénario alternatif au blocage de la réplication par un dimère est d’insérer un nucléotide en face du dimère pour poursuivre la réplication.
Ce mécanisme est connu chez les bactéries et intervient probablement chez les Eucaryotes. (Fig.7).
Figure 7 :
Le système SOS chez E-coli : (Fig.8) :
Le système SOS regroupe un ensemble de gènes (env. 30) qui est impliqué dans la réplication de l’ADN, dans la réparation de l’ADN et dans la division cellulaire et dont l’expression est contrôlée par une altération de l’ADN.
Le système SOS fonctionne comme un système de type opérateur, on se trouve face à deux états, qui utilisent ou non les protéines Rec A qui sont les protéines clé de la recombinaison procaryote :
Un état non induit, sans Rec A, durant lequel Lex A se lie aux opérateurs en réprimant la synthèse des protéines impliquées dans la réponse SOS de la cellule, les gènes ne sont donc pas exprimés.
Un état induit, avec Rec A qui est toujours présent dans la cellule mais en petites quantités.
Lors d’une altération les protéines Rec A activent leurs propres synthèses en clivant les protéines Lex A qui inhibaient jusqu’alors la transcription des gènes du système SOS dont celui de Rec A.
Les différents gènes participant au système SOS forment un régulon qui est un groupe de gènes dont l’expression est contrôlée par une même protéine.
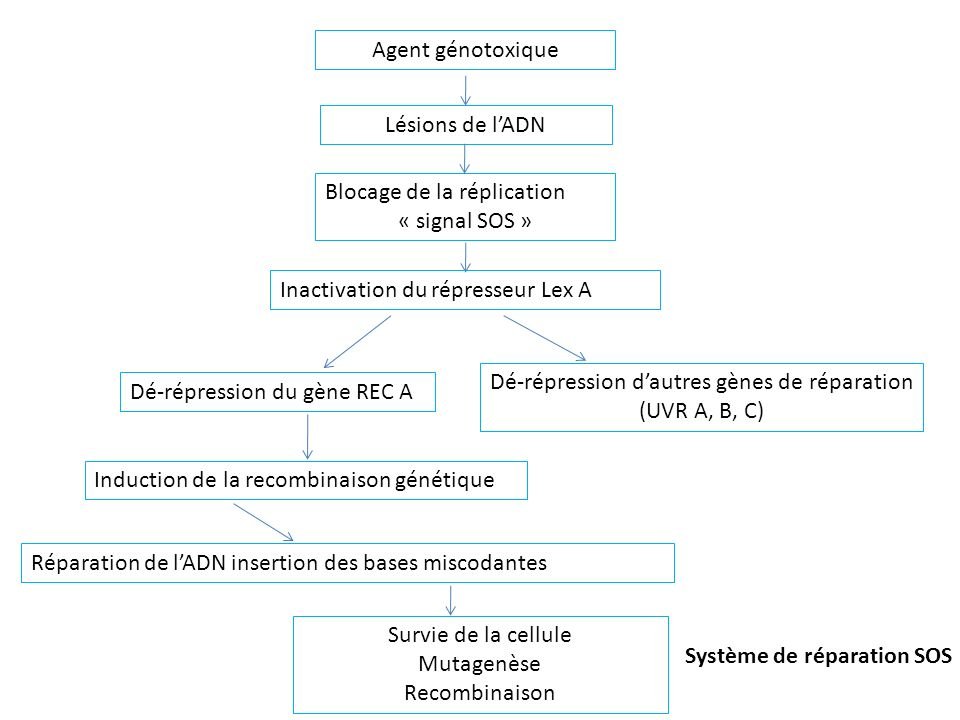

Figure 8 : Système Sos chez E. Coli
