PROTEINES
PROTEINES
Définition
Les protéines sont une classe des macromolécules de type polymère composée d’une ou plusieurs chaînes d’acides aminés (chaines polypeptidiques).
Les protéines (ou les protides) sont des éléments essentiels par leurs rôles très variés au sein d’une cellule et un organisme:
Un rôle structurel (l’actine),
Un rôle catalytique (les enzymes),
Un rôle de régulation de l’expression des gènes (les facteurs de transcription),
Caractéristiques des protéines
Chaque protéine a une structure qui est déterminée génétiquement et donc, possède une taille prédéfinie.
Dans la cellule, chaque protéine joue un rôle particulier.
Les protéines sont synthétisées et dégradées en permanence dans les cellules (équilibre permanent).
Une protéine est :
Monomérique = une seule chaîne peptidique
Multimérique = plusieurs chaînes peptidiques. Homomultimèrique = plusieurs chaînes peptidiques identiques Hétéromultimèrique = plusieurs chaînes peptidiques différentes.
Une holoprotéine ne fournit que des acides aminés, après hydrolyse.
Une hétéroprotéine fournit des acides aminés et d’autres molécules différentes, après hydrolyse.
La partie protéique: apoprotéine
La partie non protéique: groupement prosthétique
Les protéines peuvent être classées selon leur forme globale : Les protéines globulaires: myoglobine
Les protéines fibreuses : fonctions structurales ou protectrices (kératine, collagène …)
Les protéines peuvent être covalement liées à d’autres molécules: À un lipide; on parle de lipoprotéine,
À un glucide; on parle de glycoprotéine
Si c’est à un métal; on parle de métalloprotéine
Structure tridimensionnelle des protéines
Les protéines diffèrent les unes des autres parce qu’elles ont un nombre distinct et une séquence distincte de résidus d’acides aminés.
Une séquence donnée d’acides aminés s’enroule en une structure tridimensionnelle unique et complexe désigné sous le terme de conformation;
Cette conformation est réalisée grâce à l’établissement de liaisons faibles.
Cette conformation est classée par ordre de complexité croissante en structure primaire, secondaire, tertiaire et quaternaire.

La structure tridimensionnelle des protéines renseigne sur le rôle qu’elles jouent dans la cellule (relation structure-activité).
La structure primaire est la structure chimique (covalente): quels acides aminés et dans quel ordre.
La structure secondaire correspond aux structures spatiales régulières (hélices α, feuillets β, coudes β).
La structure tertiaire concerne l’arrangement dans l’espace de ces structures secondaires.
La structure quaternaire est une association de structures tertiaires: certaines protéines existent sous forme de complexes comportant alors plusieurs sous-unités (exemple: l’hémoglobine).
Structure primaire
Décrit la séquence ou l’ordre d’enchaînement des acides aminés qui constituent la protéine.
Cette séquence est fixée et traduit l’information contenue dans le gène qui code cette protéine.
Les AA sont numérotés en allant du N-terminal vers le C-terminal.
La structure primaire s’écrit en utilisant le code à 1 lettre ou le code à 3 lettres.
1 – 2 – 3 – 4 – 5 – 6 – 7 – 8 – 9 – 10 – 11- 12 -13 -14 -15 …
M – V – H – L – T – P – E – E – K – S – A- V – T – A – L …
Met-Val- His- Leu-Thr-Pro-Glu –Glu-Lys-Ser-Ala-Val-Thr-Ala-Leu … N-term C-term
Structure secondaire
1er stade de l’organisation dans l’espace d’une chaîne peptidique.
Une chaine d’AA possède au niveau des liaisons peptidiques de nombreux groupements –CO- et –NH- ceux-ci peuvent établir en eux dans l’espace des liaisons hydrogènes et former une structure secondaire.
Les structures secondaires (stables) les plus fréquentes sont l’hélice α, le feuillet plissé β et les coudes β.


L’hélice α
La chaîne principale s’enroule en spirale, vers la
droite.
La structure est stabilisée par des liaisons hydrogènes
intramoléculaires entre les résidus n et n+4.
L’hélice α s’élève de 0,54nm à chaque tour.
Elle compte 3,6 résidus par tour. .
Les plans des liaisons peptidiques sont parallèles à l’axe de l’hélice.
Les chaînes latérales R et liaisons C-H pointent vers l’extérieur.
La kératine de nos cheveux est une protéine en hélice α qui forme une fibre allongée.
| Feuillet plissé βLa chaine peptidique se trouve sous forme en zigzag. La chaîne principale est étirée et deux segments de la protéine se placent côte à côte, unis par des liaisons hydrogènes entre les groupements C=O et NH.Si les segments sont orientés dans le même sens, on parle de feuillets parallèles.Si les segments sont orientés dans le sens contraire, on parle de feuillets antiparallèles.Les chaînes latérales, R, se dressent au sommet des arêtes.La fibroïne est une protéine sécrétée par le ver à soie qui donnera le fil de soie. Cette protéine est constituée essentiellement de feuillets plissés β. | |
| Le coude β ou tour βun coude serré impliquant 4 résidus et qui permet à la chaîne de changer de direction.La chaîne principale de la protéine fait un tour en U; retrouvé souvent à la jonction de deux segments de la chaîne formant un feuillet β antiparallèle.Ces coudes contiennent en général une glycine ou uneproline. |
Structure tertiaire
La structure tertiaire consiste en une organisation des structures secondaires entre elles.
Cela implique l’apparition de liaisons hydrogènes, ioniques, de forces hydrophobes et parfois de ponts disulfure.
La structure tertiaire correspond à la structure tridimensionnelle de la protéine.
Une structure tertiaire n’est pas une structure figée : elle peut se modifier (se tordre, se déformer) sous l’effet de la fixation d’une molécule (ligand) ou sous l’effet de la variation d’un paramètre physico-chimique (pH, température).
Structure tertiaire : une protéine soluble (qui sera au contact de l’eau) va se replier de façon à ce que les résidus les plus polaires soient au contact du solvant.
Les résidus apolaires, eux, seront au cœur de la protéine de façon à ne pas interagir avec l’eau.
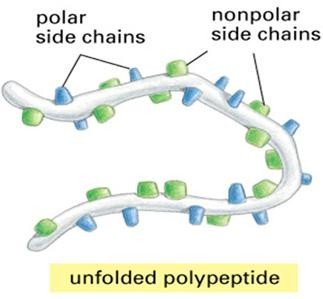
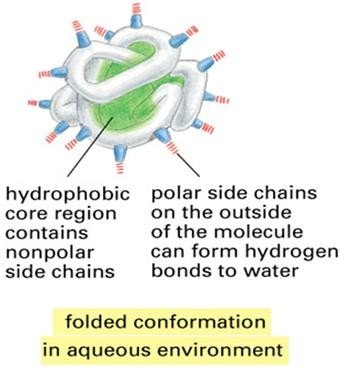
Une protéine hydrophobe (qui sera insérée dans des lipides) va se replier de façon à ce que les résidus les plus hydrophobes soient au contact des lipides qui l’entourent.
Les résidus polaires, eux, seront au cœur de la protéine de façon à ne pas interagir avec ces lipides.
Structure quaternaire
C’est l’association de plusieurs chaînes peptidiques pour donner un complexe stable et actif.
Plusieurs sous-unités tridimensionnelles (structures tertiaires) s’assemblent pour former des unités fonctionnelles beaucoup plus grandes.
Les chaînes qui constituent ce complexe sont des protomères ou sous-unités, chacune ayant une structure tertiaire définie.
L’association des différentes chaînes se fait via des liaisons faibles et parfois des ponts disulfures.
Exemple
L’hémoglobine
Un transporteur d’oxygène, Possède une structure quaternaire,
Formée de quatre sous-unités identiques 2 à 2
(2 α et 2 β).
Structure 3D de l’hémoglobine
Les différents types de liaisons ou forces impliquées dans la structuration des protéines
Structure primaire: liaisons peptidiques (covalentes), les ponts disulfure Structures II, III et IVaires : liaisons faibles éventuellement covalentes
– Liaison hydrogène se forme lorsqu’un atome d’hydrogène déjà lié par covalence à un autre atome électronégatif subit l’attraction d’un autre atome électronégatif.
Dans les cellules, les atomes électronégatifs qui participent à des liaisons hydrogène sont le plus souvent l’oxygène et l’azote.


| – Liaison ioniqueUn atome cède un ou plusieurs électrons pour former un ion chargé positivement (cation). Un autre atome capte ces électrons pour former un ion chargé négativement (anion).Il y a donc transfert d’électrons entre les atomes. (oxydation : l’atome perd des électrons et réduction : l’atome gagne des électrons).Les cations et les anions s’attirent l’un à l’autre dans une liaison ionique (En raison de leurs charges opposées).Par exemple, le chlorure de sodium (NaCl). |
| – Liaison hydrophobeComme les molécules non polaires ne peuvent pas réagir avec l’eau.Elles tendent à créer entre elles des liaisons de type interactions hydrophobes |
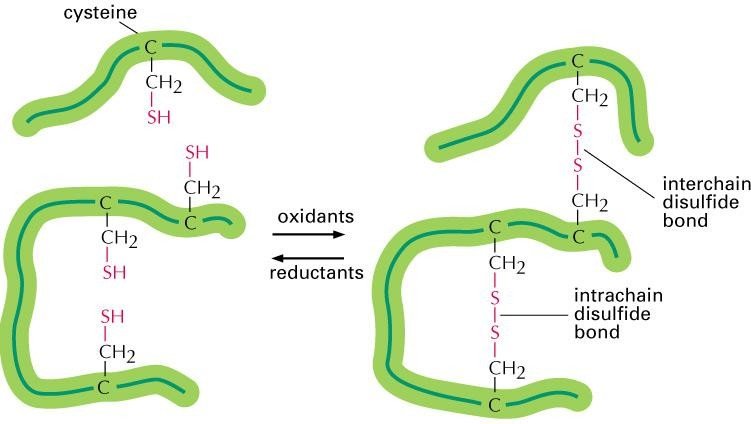
| – Les ponts disulfuresIls se forment entre deux chaines peptidiques (inter chaines) ou au sein d’une même chaine (intra chaine). |
| Schéma récapitulatif des différents liaisons faibles retrouvées dans lastructure des protéines |
Quelques différences entre les deux classes des protéines
| Protéines fibreuses | Protéines globulaires |
| Forme allongée et mince | Forme sphérique et compacte |
| Insolubles dans la cellule | Solubles dans le cytoplasme ou dans la phase lipidiquedes membranes cellulaires |
| Fonction mécanique et structurale | Agents principaux de l’activité biologique de la cellule |
| α- kératinesfibroïnecollagèneélastine | enzymes (catalyseur cellulaires)transporteurs d’oxygène ou de lipideshormonesrécepteurs intégrés à la membrane et médiateurs d’effets hormonauxImmunoglobulines (anticorps) |
Exemples de protéines fibreuses
| CollagèneProtéine extracellulaire insolubles très résistantes. 3 types: I (90%), II, III.Retrouvé partout dans l’organisme dans l’os, le cartilage, les tendons, les ligaments, les vaisseaux, etc. Structure en triple hélice α1/3 des résidus d’AA= glycineStructure tri peptidique répétée dont le premier acide aminé est toujours la glycine (Gly-X-Y).Présence d’hydroxyproline et d’hydroxylysine. Contient des sucres (glucose, galactose). |
| kératineProtéine insoluble dans l’eau; retrouvée dans la peau. Cheveux : constituée de 14 % de cystéine (ponts disulfures) = rigidité.2 types:La kératine α: formée d’hélice α = mammifères (cheveux et ongles).La kératine β: formée de feuillet β plissés antiparallèles =oiseaux (plumes) |
Techniques d’étude des structures des protéines
Les méthodes les plus importantes pour la détermination des 4 types de structures des protéines sont:
| Structure | Méthode |
| Primaire | Séquençage |
| Secondaire | Résonnance Magnétique Nucléaire, Diffraction des Rayons X |
| Tertiaire | Résonnance Magnétique Nucléaire, Diffraction des Rayons X |
| Quaternaire | Résonnance Magnétique Nucléaire, Diffraction des Rayons X |
Propriétés physico-chimiques des protéines
Masses molaires
| La masse moléculaire s’échelonne de 10KDa à plusieurs millions.La masse moléculaire d’une protéine est souvent utilisée comme élément caractéristique servant à la définition ou à la nommer:exp : P47 c’est une protéine de 47KDa | Protéine | Masse moléculaire (dalton) |
| Cytochrome c | 12300 | |
| Myoglobine | 17200 | |
| Anhydrase carbonique | 30000 | |
| Ovalbumine | 42700 | |
| Albumine | 66250 | |
| Ovotransferrine | 76-78000 |
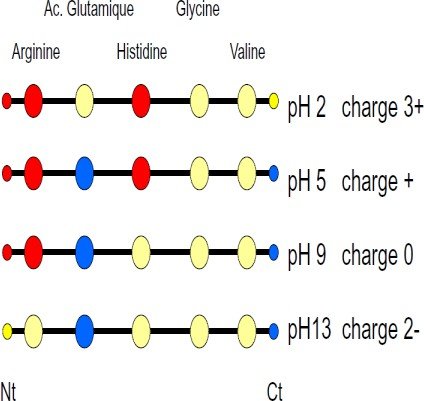
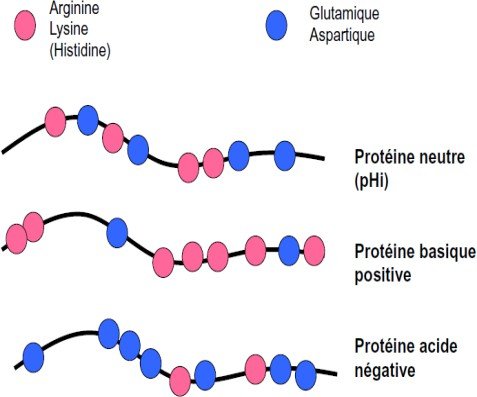
Caractère amphotère
Caractère amphotère (comme AA), avec un degré de complexité plus élevé en raison du plus grand nombre de charge mis en jeu.
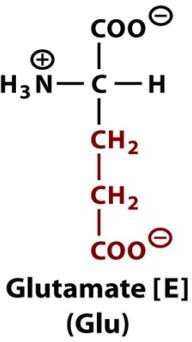
Histidine: pouvoir tampon à pH neutre avec pHi = 7,60


Solubilité
La solubilité d’un composé est la quantité maximale du composé qui peut se dissoudre dans un litre de solvant considéré.
La solubilité des protéines dépend de certains paramètres :
Influence de la concentration en électrolytes de la solution.
Influence du PH.
Influence des solvants organiques: les alcools méthyliques, l’acétone précipitent les protéines.
La solubilité est minimum au pHi et elle augmente avec la force ionique.
Stabilité thermique
Froid ou chaleur provoquent la dénaturation des protéines.
Dénaturation: modification de la structure tridimensionnelle sans modification de la structure primaire.
Perte d’activité biologique, modification des propriétés physico-chimiques.
Autres propriétés
Visible: les holoprotéines sont incolores Absorption de la lumière en UV:
Absorption à 200 nm (liaison peptidique)
AA aromatiques (absorption à 280 nm)
Coloration par fixation des colorants:
Les protéines fixent des colorants (Rouge Ponceau, noir d’amide, Bleu de coomasie…)
Coloration par réaction: permet le dosage des protéines : Réaction au biuret et Lowry
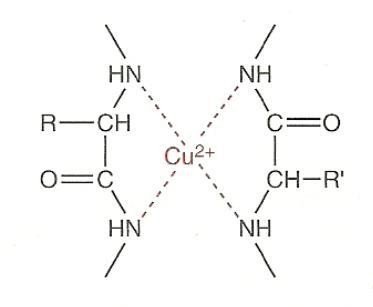
Réaction au biuret
La réaction caractérise la liaison peptidique ; elle s’applique donc à l’analyse des protéines et des peptides (à condition que ces derniers comportant au moins quatre acides aminés afin d’obtenir trois liaisons peptidiques).
En milieu alcalin, les protéines qui possèdent au moins 3 liaisons peptidiques forment avec les ions cuivre II (Cu2+) un complexe bleu-violet à 540 nm dont l’intensité de la couleur est proportionnelle à la concentration en protéines.
Détermination de la structure primaire des protéines
Stratégie générale
La détermination de la séquence complète en AA d’une protéine et l’ordre de ces AA passe par les étapes suivantes:
Extraire, séparer et purifier la protéine.
Rompre les ponts disulfures (sous unités), fragmenter et hydrolyser la protéine (analyse = composition en Aa).
Séquençage de la protéine et identification des Aa aux extrémités Ct et Nt.
Réarrangement de la séquence de la protéine.
Techniques de séparation et de purification
Les diverses techniques pour séparer les polypeptides se basent sur la taille, la solubilité dans un solvant particulier, la charge ou l’aptitude de ces polypeptides à se lier à un support.
| Paramètre | Technique |
| Densité | Ultracentrifugation |
| Solubilité | Précipitation au sulfate d’ammonium |
| Taille | Filtration sur gel |
| Charge | Chromatographie d’échange d’ions, électrophorèse, isofocalisation. |
| Affinité | Chromatographie d’affinité |
Ultracentrifugation
Procédé de séparation des composés d’un mélange en fonction de leur différence de densité en les soumettant à une force centrifuge.
L’appareil utilisé est une machine tournante à grande vitesse appelée centrifugeuse.
Chromatographie d’exclusion
Ou chromatographie sur gel filtration: la séparation est basée sur la taille des protéines.
Le gel est composé de billes trouées avec des trous de différents diamètres; les petites molécules pénètrent dans les trous et sortent tardivement et les grandes sortent les premières.

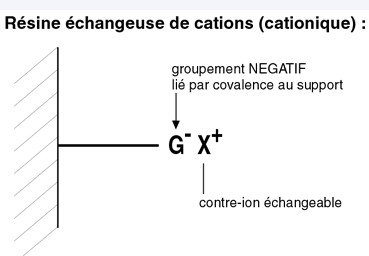
Chromatographie sur résine échangeuse d’ions
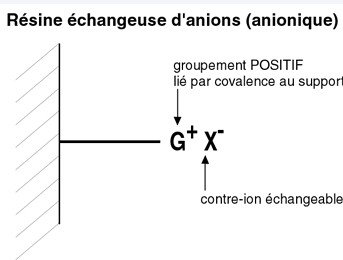
C’est un procédé basé sur la charge des protéines.
Le support solide (résine) contient des ions chargés positivement (résine échangeuse d’anions
= diéthylaminoéthylcellulose ou DAEA cellulose) ou négativement (résine échangeuse de cations
= carboxyméthylcellulose ou CM).
Les protéines se lient à ces groupements selon leur charge.
Puis il ya récupération de ces protéines par interruption de ces liaisons.
| Chromatographie d’affinitéNécessite la reconnaissance de la protéine par un ligand porté par la phase solide ;Méthode plus efficace que la chromatographie par échange d’ions ou la chromatographie par gel filtration.Condition: il faut avoir un ligand pour la protéine recherchée. |
Electrophorèse en gel de polyacrylamide (PAGE) avec SDS
Le Sodium dodécylsulfate (SDS), dénature les protéines (séparer les sous unités et les rendre sous une structure primaire) et leur attribue une même charge négative.
La séparation dans le PAGE avec SDS est fonction de la masse molaire car toutes les molécules sont chargées de la même façon.
Focalisation isoélectrique : Séparation basée sur le pHi des protéines.
On utilise un gradient de pH et on dépose le mélange protéique.
En appliquant un champ électrique, les protéines vont être séparées dans la solution quand le pH du milieu s’accorde à leur pHi.
Electrophorèse sur gel d’agarose et acétate de cellulose
L’électrophorèse est une méthode physique de fractionnement des molécules ionisées.
En solution, les protéines acquièrent une charge électrique.
Si on les met dans un champ électrique, elles se déplacent et le sens de migration dépend, dans ce cas là, du pH de la solution étudiée par rapport au pHi des protéines (migration vers l’électrode de signe opposé à leur charge).
Ainsi, les protéines sont séparées en 6 fractions : albumine et 5 groupes de globulines (α1, α2, β et γ globulines)

+
_


Fragmentation
Détermination de la composition en acides aminés
C’est l’identification des acides aminés constitutifs d’une protéine ou d’un peptide
Cette étape comporte :
La rupture de la séquence peptidique par hydrolyse (chimique ou enzymatique) des liaisons peptidiques.
L’analyse qualitative et quantitative des acides aminés du mélange obtenu après hydrolyse.
Rupture des ponts disulfures
Permet la séparation des chaînes polypeptidiques liées par des ponts disulfure par le mercapto-éthanol
Empêche la conformation native qui pourrait résister à l’action des agents protéolytiques
Coupures intra-chaine peptidique
Traitements chimiques et enzymatiques (endopeptidases).
Séquençage
Détermination des extrémités C-terminal et N-terminal par voie enzymatique (exopeptidases ou carboxypeptidases).
Détermination du C-terminal par méthode chimique
Hydrazinolyse
L’hydrazine à 100°C attaque toutes les liaisons peptidiques et donne des dérivés hydrazides d’acide sauf pour l’acide aminé C-terminal qui reste normal.
Détermination du N-terminal par méthodes chimiques
Méthode de Sanger (FDNB) Méthode de dansylation Méthode récurrente d’Edman
Détermination de la séquence en AA
C’est l’établissement de l’ordre dans lequel les AA sont liés.
Les fragments protéolytiques sont séparés par HPLC et séquencer ainsi l’un après l’autre par la méthode d’Edman après identification des extrémités C et N terminales.
a séquence du polypeptide original est obtenue en comparant les séquences en AA d’une série de fragments peptidiques avec celles d’une deuxième série dont les sites d’hydrolyse recouvrent ceux de la première série:

