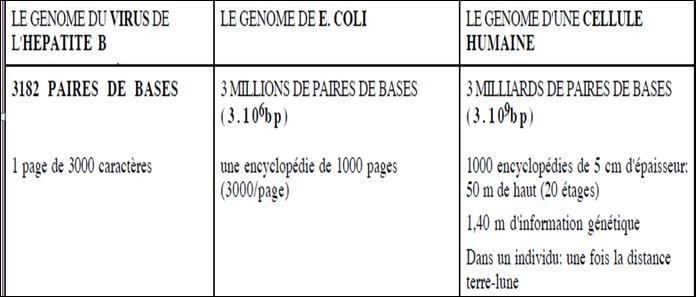
Biologie moléculaire
Biologie moléculaire
Science consacrée à :
l’étude des molécules porteuses du message héréditaire (ADN, ARN) (structure du génome),
La synthèse des protéines , altérations (mutations) chez les organismes vivants.
La biologie moléculaire permet d’analyser, à travers les molécules, et les altérations du génome = mutations.
La biologie moléculaire s’intéresse également au mécanisme de
l’expression des gènes que celui-ci soit normal ou pathologique.
La biologie moléculaire désigne également les techniques d’étude des gènes PCR , clonage, hybridation moléculaire, séquençage, puce à ADN ( DNA Chips).
ACIDES NUCLEIQUES
INTRODUCTION
Les acides nucléiques ont été caractérisés chimiquement au début du 20ème siècle même si leur rôle est resté relativement longtemps inexpliqué.
Les acides nucléiques ont été isolés initialement des noyaux des
cellules.
On distingue deux grands types:
les acides désoxyribonucléiques (ADN) localisés dans le noyau des cellules
les acides ribonucléiques (ARN)localisés dans le cytoplasme
cellulaire.
Les acides nucléiques (ADN et ARN) sont des macromolécules et
comportent des sous-unités appelées nucléotides.
Ils jouent également un rôle fondamental dans le métabolisme sous forme di- et tri-phosphorylée ainsi que dans la transmission de l’information dans la cellule (AMPc et GMPc).

4


5
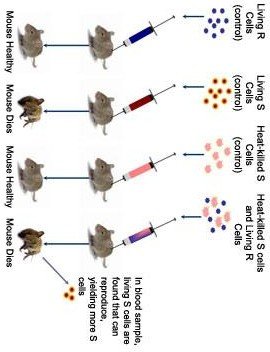
Frederick Griffith
Griffith’s Experiment : 1928
ACIDES NUCLEIQUES

1877 – 1955
INTRODUCTION
Expérience d’Oswald Avery (1944)
Souche R + protéines de S
Souche R + capsule de S Souche R + lipides de S
Souche R + acides nucléiques de S
Souche R + ac.nucléiq. de S + DNase
Souche R + ac.nucléiq. de S + RNase
→ADN support chimique de l’hérédité 7
– Elle permet de savoir quel composant viral : A.N ou protéine, pénètre la cellule pour servir de plan pour la reproduction
virale.
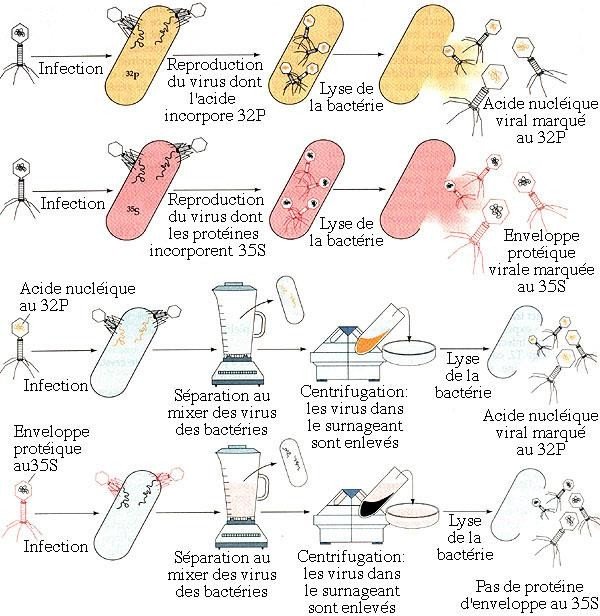

Expérience de HERSHEY et CHASE, 1952
On cultive d’une part des virus bactériophages T2 sur des bactéries elles-mêmes élevées sur un milieu contenant du soufre sous sa forme isotopique 35 S*
Les protéines virales formées, qui contiennent du soufre, sont marquées R**
En réalisant d’autre part une culture semblable sur un milieu où le phosphore est présent sous la forme de l’isotope radioactif 32P, on marque distinctement les acides nucléiques viraux.
Après avoir séparé, dans chacune des 2 cultures, les virus, tous marqués, des bactéries qui les ont reproduits, on infeste simultanément deux nouvelles cultures bactériennes, chacune avec une souche différente des phages marqués
On constate, en analysant les culots bactériens grâce aux traceurs
8

9

10

1953
11
UNITÉ de base de l’ADN = LE NUCLÉOTIDE
Les polynucléotides biologiques sont :
le support moléculaire de l’information génétique : l’ADN (et ARN pour certains virus) est
le support de l’hérédité et du codage des composés biologiques (les ARN, les protéines)
des effecteurs de l’expression de l’ADN en peptides et protéines : acide ribonucléique
dont l’abréviation est ARN (RNA : anglo-saxon) regroupés en trois classes :
les ARN messagers (ARNm)
les ARN de transfert (ARNt)
les ARN ribosomaux (ARNr)
UNITÉ de base de l’ADN = LE NUCLÉOTIDE
Sucre : désoxyribose
Groupement phosphate
Base azotée
Un nucléotide comporte trois composants:
de l’acide phosphorique + un ose + une base.
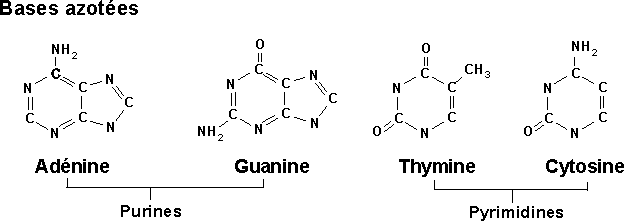
A- BASES AZOTÉES
Il y a 4 sortes de bases azotées:qui appartiennent à deux classes de molécules selon le noyau aromatique qui en constitue le squelette.

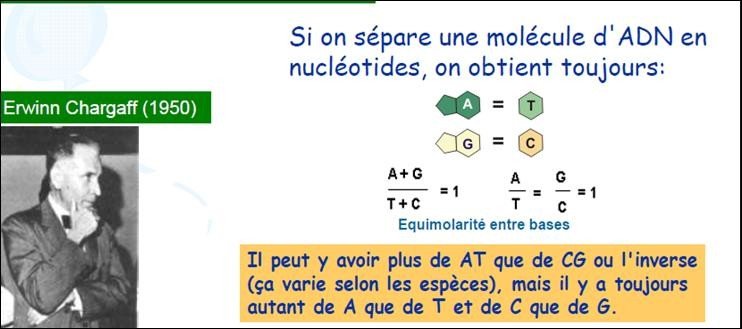
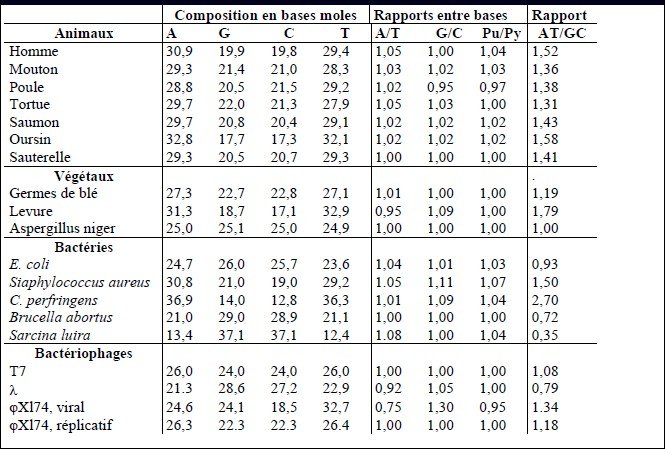
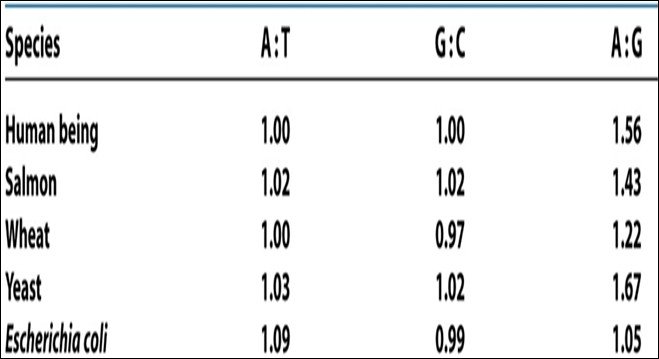
Il peut y avoir plus de AT que de CG ou l’inverse (ça varie selon les espèces), mais il y a toujours autant de A que de T et de C que de G.
A = T et C = G
2 sortes de bases azotées hétérocycliques
bases puriques (noyau purine)
bases pyrimidiques
(noyau pyrimidine)
4
3 N
5
2
6
N
1
6 7
5
N
N
N
4
N
1
8
2
3 9
Le noyau pyrimidine est le plus simple : c’est un noyau aromatique hexagonal à six atomes, quatre carbones et deux azotes (n° 1 et 3).
1- Bases pyrimidiques
fonction amide
O
NH2
N
3
2
4
H
O
1
N H
5
6
H
fonction imino-amine
HN 4
3 5
CH3
2 1 6
O N H
H
cytosine (C) thymine (T)
5-méthyl-uracile
uracile (U)
ADN/ARN ADN ARN

HN
3
2
4
H
O
1
N H
5
6
H
fonction amide
O
Les bases pyrimidiques sont au nombre de 3 : la cytosine, la thymine et l’uracile
La cytosine :le carbone 4 est substitué par une fonction amine et le carbone 2 par une fonction cétone.
L’uracile : les carbones 2 et 4 portent des fonctions cétone.
Bases pyrimidiques
NH2
O
O
HN
H
3
2
4
O
1
N H
5
6
H
fonction amide
fonction amide
fonction imino-amine H
N 4
3 5
HN 4
3 5
CH3
2 1 6
O N H O
H
2 1 6
N H
H
cytosine (C) thymine (T)
5-méthyl-uracile
uracile (U)
ADN/ARN ADN ARN
La désamination oxydative de la cytosine en uracile:
Dans l’ADN, l’uracile sera réparé par des enzymes de réparation
Dans l’ARN, ces changements ne sont pas graves car la durée de vie de l’ARN et des protéines est courte.
L’absence de T permet la reconnaissance de l’ARN pour les enzymes de dégradation et un gain d’énergie.
La désamination oxydative de la cytosine Methylée → Thymine (pas de réparation) : Dans L’ADN, elle est source de mutations (Pts chauds de mutations ou hot spot fréquen1t7e dans les ilots CG)
Bases pyrimidiques
Tautomérie : équilibre impliquant la migration de protons
NH2



NH2
H-N
3
2
4
O
5
1 6
N
+
H
3
N
4
3 2 5
2 1 6
H H
O N H H
1
H
Echange d’hydrogène entre N1 et N3
Cet équilibre ne concernera pas les bases où N1 est substitué.
Bases pyrimidiques
Équilibre amino-imino (cytosine)
H
NH2
N
3
2
4
1
N
H
5
6
NH
HN
3
2
4
H
O
1
N
H
5
6
H
O H
99,99% 0,01%
En solution ces 2 espèces moléculaires sont en équilibre (équilibre fortement déplacé vers la forme amino).
Bases pyrimidiques
Équilibre énol-cétone (thymine-uracile)
H


5
1 6
N H
4
3
2
N
O
H+
1
2
H-N
H
3
2
4
O
1
N H
5
6
H
O
O O
N
3
2
4
5
1 6
N H
H H
H O H
H
En solution forme cétone prédominante
Bases Pyrimidiques Modifiées
Il existe des bases pyrimidiques modifiées (chez les phages par
exemple…).
NH2
4
3 5
CH3
NH2
4
3 5
CH2OH
2 1 6
N H
H
2 1 6
N H
H
5-hydroxyméthylcytosine
5-méthylcytosine
Methylation de l’ADN : 70 à 80 % des cytosines de dinucléotides CpG peuvent être méthylés chez les mammifères.
produite par oxydation de la 5-méthylcytosine
observée pour la 1ère fois chez des bactériophages
puis a été observée en abondance dans le cerveau chez les humains les souris et dans les cellules souches embryonnaires.
Elle joue un rôle important en épigénétique 22
Bases Pyrimidiques Modifiées
Des analogues de l’uracile (5-halogéno-uracile) sont utilisés en thérapeutique anti-tumorale (Anticancéreux).
.
O
HN
X
3
2
4
O
1
N H
5
6
H
X = F, Cl, Br, I
2 – Bases puriques
bases pyrimidiques (noyau pyrimidine)
bases puriques
(noyau purine)
6
5
1 N
7
N
8
2
N
3
4
N
9
4
3 5
N
N
2 6
1
Les purines ont un double noyau aromatique comportant :
à gauche : un cycle hexagonal de 4 carbones et 2 azotes
à droite : un cycle pentagonal de 3 carbones (dont 2 communs avec le précédent)
BASES PURIQUES
O
NH2
N
6
1
2
5
4
3
N
N
7
8
9
NH
HN 6 N
7
1 5
2 4 8
9
H2N N NH
3
adénine (A)
ADN/ARN
guanine (G)
ADN/ARN
Les bases puriques sont au nombre de 2 : l’adénine et la guanine.
L’adénine : le carbone 6 est substitué par une fonction amine.
Elle est la seule des bases nucléiques dont la formule ne contient pas d’atome d’oxygène.
La guanine :le carbone 2 est substitué par une fonction amine et le carbone 6 par une
fonction cétone.
BASES PURIQUES
D’autres purines existent : xanthine, hypoxanthine, acide urique, caféine…
O O
HN
N
O
N H
NH
N
N
N H
NH
xanthine hypoxanthine
Intermédiaire du métabolisme de l’adénine et de la guanine
O CH3
N
BASES PURIQUES
O O
HN
O N
CH3
CH3
N
N
O N
N CH3 N NH
O N N
Théobromine ( Cacao du chocolat)
CH3
CH3
CH3
théophylline
caféine


BASES PURIQUES
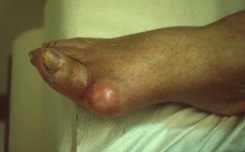
– L’acide urique une molécule
quasiment insoluble dans l’eau résultant de la dégradation et de l’excrétion des purines.
Une augmentation de sa concentration dans le sérum sanguin, appelée hyperuricémie, entraîne la goutte, responsable d’arthropathies et de lithiase rénale par formation de cristaux dans les reins, appelés calculs rénaux.
(goutte)
O
HN
NH
O
O
NH
O
NH
HN
N
O
–
O
NH
NH
(goutte)
ACIDES NUCLEIQUES
2- Le Pentose = deux sortes de sucres
D-ribose
D-désoxyribose
(2’-désoxyribose)
5’
HOH2C
O
4’ 1’
3’ 2’
5’
OH HOH2C
OH
4’ 1’
3’ 2’
ARN ADN
OH OH OH H
pentoses sous forme furanique (5 atomes dans le cycle)
Le désoxyribose, est dérivé du ribose par une réduction de la fonction alcool
secondaire du carbone n°2 qui confère à l’Acide Nucléique une plus grande stabilité
ACIDES NUCLEIQUES
Liaison base-sucre nucléoside
N
N 4 N 6 7
3 5 1 5
2 6 2 4 8
1 3 9
N
5’ H
H2O
N
5’ H
H2O
HOH2C
OH
4’ 1’
3’ 2’
HOH2C
O OH
4’ 1’
3’ 2’
OH OH / H
OH OH / H
Les sucres (ribose ou désoxyribose) se lient aux bases azotées par des liaisons impliquant un des azotes de la base (azote n°1 des pyrimidines ou azote n°9 des purines) et le carbone n°1
NUCLÉOSIDES ATYPIQUES
AZT: zidovudine: Retrovir
31
ATB
ACIDES NUCLEIQUES
3 – acide phosphorique : H3PO4
O
P
H-O O-H
O-H
Les différentes fonctions acides ont des pKa variables.
L’acide phosphorique (H3PO4) possède trois fonctions acide.
Deux de ces fonctions sont estérifiées dans les ADN et les ARN.
La troisième fonction acide est libre.
L’H3PO4 permet la solubilisation de l’ADN dans l’eau grâce à leurs charges (-)
Il est responsable de la fonction acides des acides nucléiques.
Les H3PO4 permettent la polymérisation des acides nucléiques
(nucléotides).
32
ACIDES NUCLEIQUES
Liaison acide phosphorique-sucre= Estérification
OH
O P OH O
–
5’
HO-H2C
O OH
4’ 1’
3’ 2’
OH
O P O O
H2O
5’
CH2
–
OH OH / H
O OH
4’ 1’
3’ 2’
OH OH / H
ACIDES NUCLEIQUES
Liaison base-sucre – acide phosphorique nucléotide
nucléotides pyrimidiques
NH2 O
H3C
H
6
5
1
N
4
2
5’
CH2
O
3
N
O
4’ 1’
3’ 2’
4
5
3
N
6
2
5’
CH2
O
1
N
O
4’ 1’
3’ 2’
OH
O P
O
OH
O P
O
O – O –
HO H
désoxycytosine-5’-monophosphate (dCMP)
HO H
désoxythymidine-5’-monophosphate (dTMP)
La liaison d’un nucléoside avec un phosphate se fait par une estérification de la
fonction alcool Iaire (C 5’) du sucre et une des 3 fonctions acides du phosphate.
L’ester obtenu est un nucléotide = formé d’une base azotée, liée par une liaison
ACIDES NUCLEIQUES
nucléotides puriques
OH O P
O
NH2
N
8
7
6
5 1
N
4 2
5’
CH2
9
3
N
4’
O N
1’
3’ 2’
OH O P
O
N
7
8
5’ 9
CH2 O N
O
6 N
5 1
4 3 2
N
NH2
O – O –
4’ 1’
3’ 2’
HO H
désoxyadénosine-5’-monophosphate (dAMP)
HO H
désoxyguanosine-5’-monophosphate (dGMP)
La liaison d’un nucléoside avec un phosphate se fait par une estérification de la
fonction alcool 1aire (C 5’) du sucre et une des 3 fonctions acides du phosphate.
L’ester obtenu est un nucléotide = formé d’une base azotée, liée par une liaison osidique avec un sucre, lui-même lié par une liaison ester avec un phosphate.
ACIDES NUCLEIQUES
Dans les cellules les nucléotides sont retrouvés sous forme nuléosides mono, di- et
triphosphates
adénine

liaisons anhydride d’acide
liaison phosphoester
pKa = 7



O
P O
O
P O
O –
5 1
4 2
9
–
HO
NH2
O P
O
O
O
–
5’
CH2
N
8
4’
O N
1’
7
3
N
6
3’ 2’
N
HO OH
adénosine AMP ou adénylate
ADP
ATP
ACIDES NUCLEIQUES
A partir d’ATP, et grâce à une enzyme appelée adénylate cyclase, il y a synthèse d’AMP cyclique.
O O O


NH2
N
8
7
6
5
1
N
4
O
–
5’
CH2
9
3
N
2
O N
4’ 1’
3’ 2’
OH OH
NH
2
N
8
7
6
5 1
N
4 2
5’
CH2
9
3
N
O
4’
O N
1’
3’ 2’
O = P
– O
O
OH
HO P O P
O
P

–

–
O O
adénylate cyclase
O
ATP
O O
HO P O
P
OH +

–

–
O
O
PPi
AMPc (second messager)
On désigne par nucléotides les nucléosides monophosphates : AMP ou acide adénylique, dTMP ou acide désoxythymidylique, etc…
Les nucléosides polyphosphates sont des diphosphates : ADP ou GDP… ou encore des triphosphates, les plus riches en énergie : ATP ou GTP ; etc…
Les acides nucléiques sont formés par une polycondensation de nucléotides AMP,
CMP, GMP et UMP pour les acides ribonucléiques, dAMP, dCMP, dGMP et dTMP
38
pour les acides désoxyribonucléiques
La liaison phosphodiester
39
ACIDES NUCLEIQUES : NOMENCLATURES
P
5’
OH
CH3
5’ H
O
4 N H
5 3
6 2
1
O
²²Thymine (T)
5’ T
P 3’
5’ G
P 3’
5’ A
P 3’
5’pTpGpApC 3’
TGAC
O P
O
CH2 O N
O – 4’ 1’
3’ 2’
O
N 6 H
N
7 5 1
H 8 4 2
5’ HO 3’ C
Guanine (G)
TgAC
tgac
O H
C-O-P-O-C
O P O
9
5’ N N
3
H
N
CH2 O H N H
H
O – 4’ 1’
Liaison phosphodiester
3’ 2’
N 6
7 5 1
N
H 8 4 2
Adénine(A)
les nucléotides sont liés entre
eux par des liaisons ester.
O H
O P
O
9
5’ N N H
3
CH2 O H N H
L’H3PO4 présente ses deux O –
fonctions acides bloquées
dans la formation d’ester :
4’ 1’
3’ 2’
H 4 N
5 3
6 2
Cytosine (C)
liaison phosphodiester :
– a la liaison ester entre
O H
O P
O
5’ H N O
CH2 O
1
H3PO4 et l’OH en 3’ de l’ose,
– b à la liaison ester en 5’ de
O – 4’ 1’
3’ 2’
3’
40
l’ose HO H

Sens de lecture d’un acide nucléique:








Par convention, on lit toujours un acide nucléique dans le sens de l’extrémité 5’ comportant en règle un groupement phosphate) vers l’extrémité 3’ qui possède un OH libre.



3’
5’
T
A C A
G
A
T G T C
5’
41 3’
La séquence des bases d’un ADN par convention sera écrite soit dans le sens vertical ou dans le sens horizontal en précisant les extrémités 5’ et 3’ et on indique seulement les bases correspondantes (A, T, G ou C).
ACIDE DÉSOXYRIBONUCLÉIQUE
Les ADN présentent plusieurs caractéristiques propres et qui les opposent aux ARN:
L’ose: le 2’-désoxyribose (remplacé par le ribose dans les ARN).
Les bases: A, C, G et T, soit 2 bases puriques A et G et 2 bases pyrimidiques :C et G.
Dans les ARN, T est remplacé par U (uracile).
Les polymères de nucléotides: La molécule
d’ADN est constituée en règle de deux chaînes (ou brins) de nucléotides contrairement aux molécules d’ARN qui sont le plus souvent sous forme d’un seul brin.
42
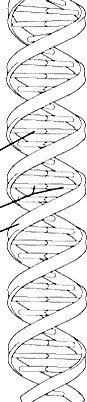
Structure de l’ADN
L’ADN est formé de deux chaînes de polynucléotides
antiparallèles (vont dans des directions opposées);
Les bases sont presque perpendiculaires à l’axe (inclinaison de 6o);
Les bases sont enfouies à l’intérieur de la structure, avec le squelette sucre-phosphate à l’extérieur;
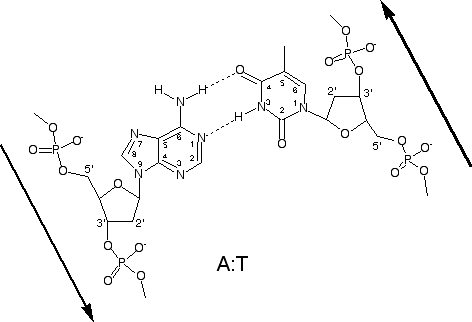
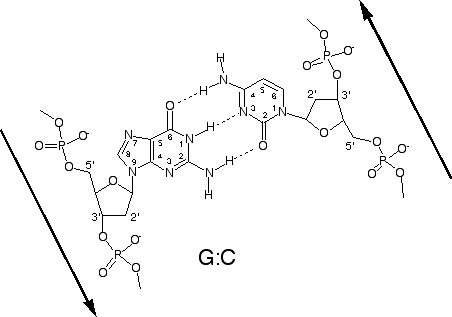
Les deux chaînes sont maintenues ensemble via la formation de ponts H entre bases azotées:
A forme 2 ponts H avec T (paire de base AT)
G forme 3 ponts H avec C (paire de base GC)
Cette relation A:T et G:C dicte la complémentarité des deux chaînes:
La nature de la base sur un brin donne immédiatement la
nature de la base sur le brin opposé;
43
Structure de l’ADN
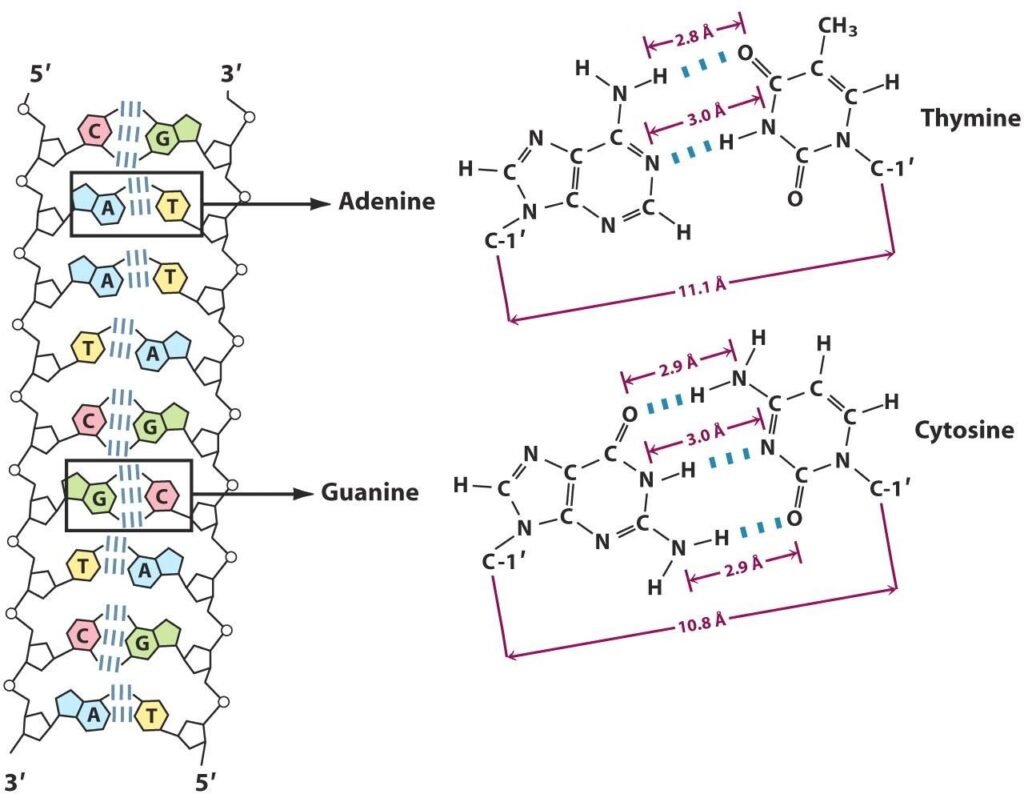
bases
Squelette sucre-phosphate 44
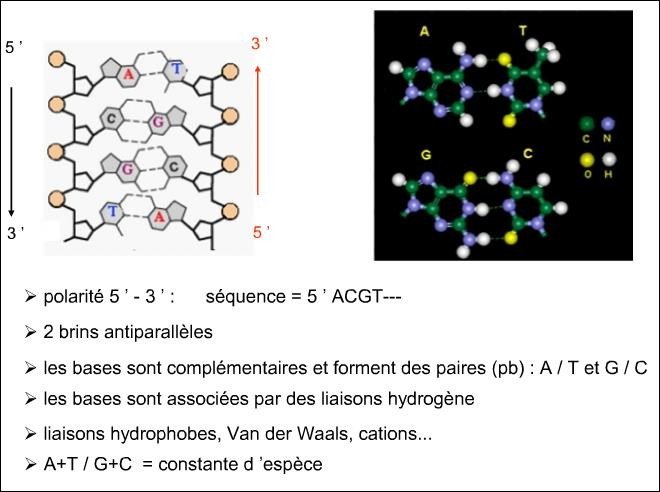
45
46
James Watson (1928- )
Francis Crick (1916-2004)



Nobel 1962
2003 : 50 ans après la découverte de la structure de l’ADN
47
La structure secondaire:
L’ADN est formé de deux chaînes antiparallèles, complémentaires et hélicoïdales.
L’ADN est bicaténaire: c’est un original, il faut une sauvegarde.
Chaque brin est le back up de
l’autre.
Structure de l’ADN
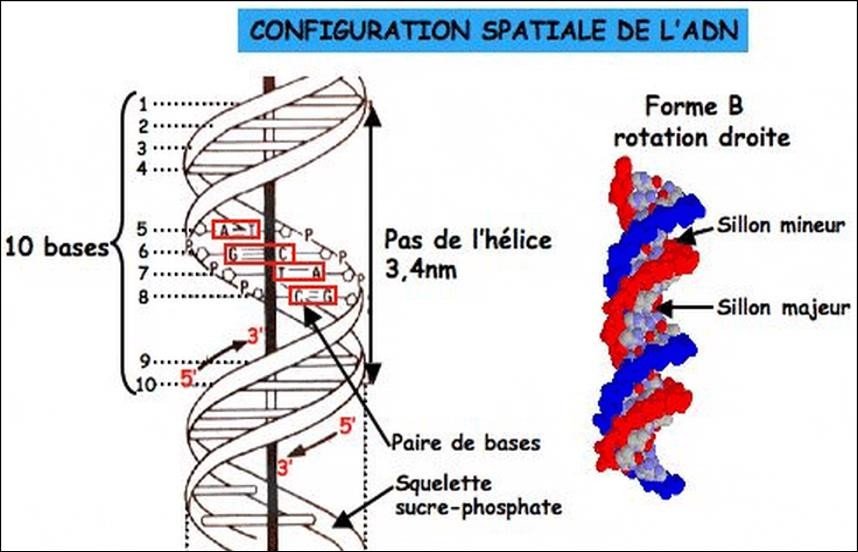
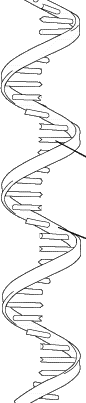
Les deux chaînes polynucléotidiques forment une hélice droite:
Environ 10 paires de bases par tour d’hélice;
3.4 Å entre 2 bases
34 Å par tour
20 Å de diamètre
Présence de deux crevasses sillons à la surface de l’hélice:
Petit sillon faible distance entre les deux chaînes;
Grand sillon: plus grand espace entre les deux chaînes;
1 Å (Ångstrom) = 0.1 nm = 1 x 10-10 m
49
Certains ATB peuvent s’insérer dans un des sillons de l’ADN : Nétropsine
50
d’autres
s’intercalent entre les
bases : Daunomycine
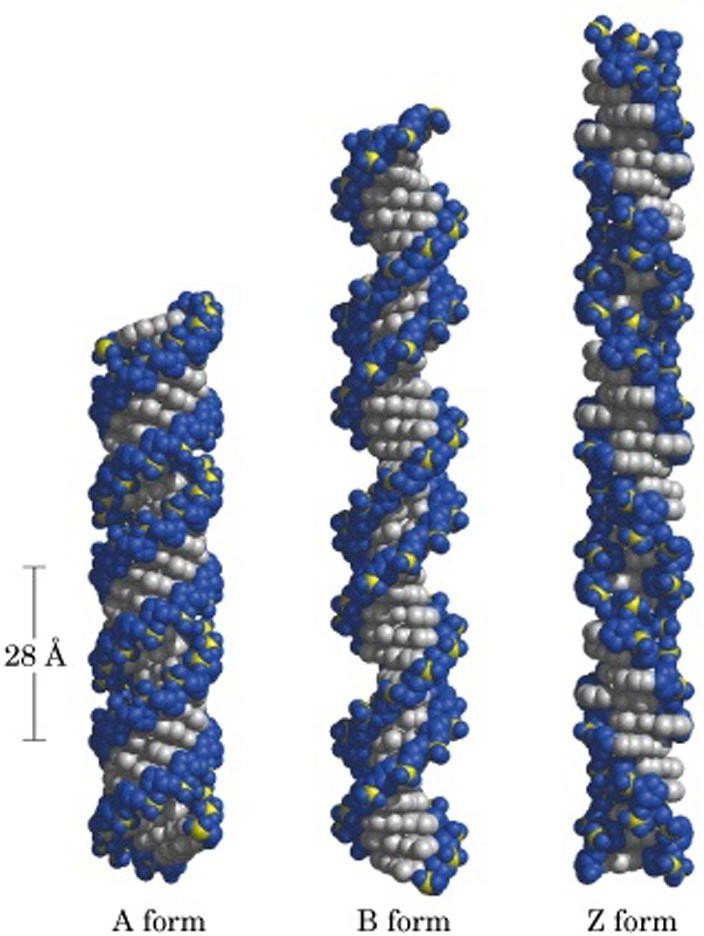
Les formes de l’ADN.
La forme B de l’ADN est la forme biologique la plus importante.
décrite en 1953 par Crick et Watson.
Les caractéristiques de l’hélice régulière sur les suivantes:
–10 paires de bases par tour de spire
– le pas de l’hélice est de 3,4 nm
–et le diamètre de l’hélice est de 2 -2,4 nm.
Dans les cellules, la forme de l’hélice est un peu plus compacte et comporte environ 10,5 paires de bases par tour de spire.
Une caractéristique importante de la forme B de l’ADN est la présence de deux types de sillons appelés sillon majeur (1,2 nm de large) et sillon mineur (0,6 nm de large).
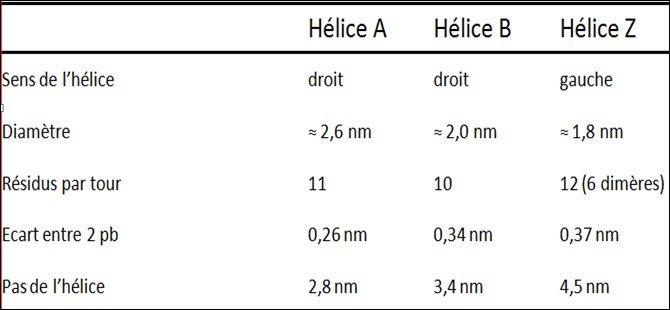
Hélice A= Duplex ARN/ARN et ADN/ARN
Plus large: 26 Å
Plus courte: 11 bp/turn
Distance par paire de base: 2,6 Å
Les bases sont plus inclinées (20o)
Rôle :
structure secondaire du RNA; complexes RNA/RNA ; hybrides RNA/DNA (réplication et transcription).
Comme pour l’ADN : les molécules hybrides ADN/ARN et les duplexes d’ARN/ARN suivent les mêmes règles de complémentarité et
d’antiparallélisme.
Cependant, le 2’OH de l’ARN affecte la structure de l’hélice.
La forme Z de l’ADN
Initialement : réaction de laboratoire (Rich, 1979) avec l’oligonucléotide
artificiel d(CGCGCG).
Actuellement, on peut obtenir du Z-DNA dans des conditions plus physiologiques.
Le Z-ADN forme une double hélice à rotation gauche avec 12 paires de
bases par tour d’hélice.
le pas d’hélice est plus important (4,5nm)
le diamètre de l’hélice est plus petit 1,8
contient seulement un sillon.
Localiation des Helices de type Z:
séquence alternée de Pu et Pyr:séquences riches en (GC)n dans le génome : îlots GC.
Peut être reconnu par des Ac anti-Z DNA.
De telles séquences peuvent se retrouver dans des opérateurs comme “lac operator” ce qui augmente l’affinité pour le lac repressor d’env. 1000X.
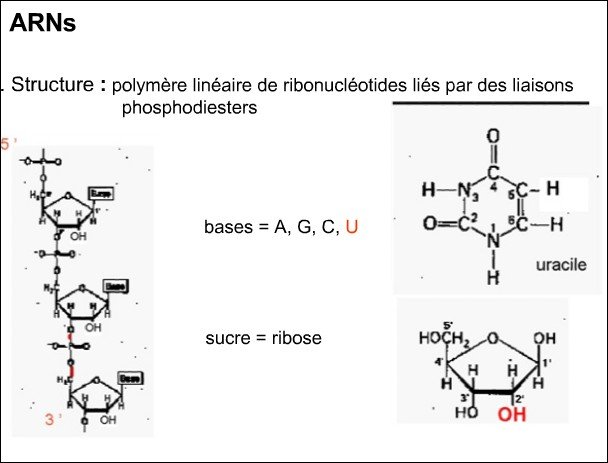
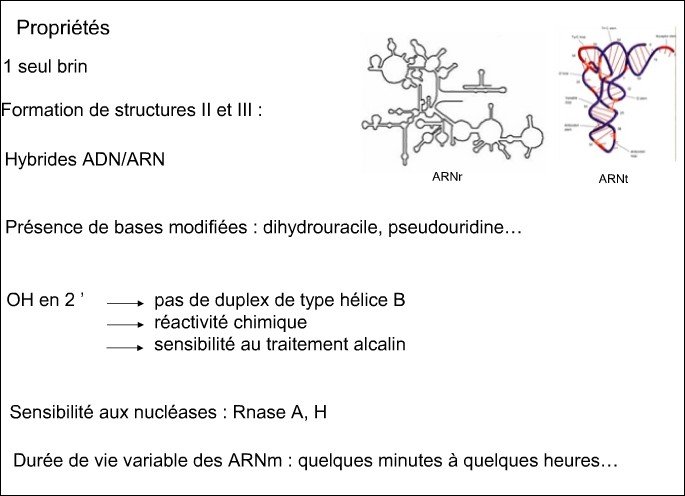
LES ARNs

Small nuclear)
gRNA : ARN génomique ( qui constitue le génome de certains virus)
RNA antisens : petit ARN complémentaire d’une portion d’un autre ARN
et inhibant sa fonction ( peuvent être naturels ou obtenus par génie génétique)
58
Propriétés physico-chimiques de l’ADN
Densité
On exploite la densité par centrifugation dans un gradient de chlorure de césium (CsCl).
Au cours de la centrifugation il se forme un gradient de chlorure de césium, l’ADN se concentre en une bande à l’endroit où la densité de la solution de CsCl est égale à la sienne.
Si on centrifuge des protéines, de l’ADN et de l’ARN on remarquera la répartition de l’ARN au fond du tube (plus dense), l’ADN au milieu et les protéines en haut (moins dense).
Ceci est du à leur différence de densité
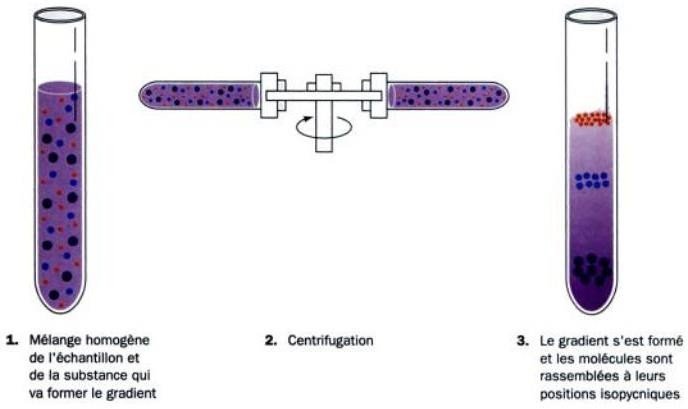
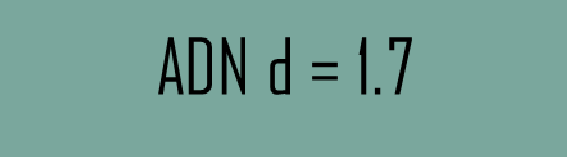

Densité des acides nucléiques
Protéines d=1.4
ARN d = 2
Poids moléculaire
Le poids moléculaire de l’ADN est très élevé.
Il est déterminé par:
Diffusion de la lumière,
Mesures de constante de sédimentation et de viscosité intrinsèque,
Microscopie électronique.
L’ADN humain fait en moyenne un PM de 6 x 1010 par chromosome.
Ce paramètre est mis à profit lors de méthodes comme la chromatographie et l’électrophorèse.
Solubilité et viscosité
La présence de groupements phosphates (OH ionisés) donne un caractère acide aux acides nucléiques.
A pH physiologique les acides nucléiques portent une charge négative, qui est uniquement due aux groupements phosphates car à ce pH les bases ne portent aucune charge.
De ce fait, les acides nucléiques sont solubles dans l’eau.
L’ADN se dissout facilement dans les solutions salines diluées et entraine une augmentation importante de la viscosité de la solution.
A forte concentration en sels l’ADN et l’ARN précipitent et peuvent être récupérés après centrifugation.
Les alcools, comme l’éthanol, précipitent également les molécules d’ADN sous forme d’agglomérat en longues fibres.
Les solutions d’ADN ont une très grande viscosité résultant de la structure longue et rigide de la double hélice.
Un ADN double brin possède une viscosité supérieure à celle d’ADN simple brin.
Propriétés spectrales Absorption dans l’UV
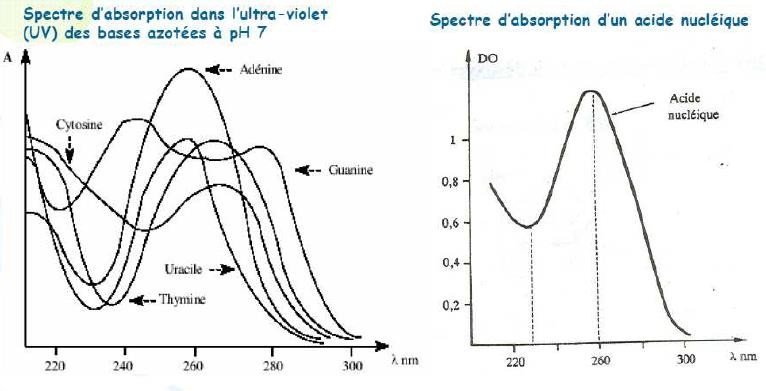
Bases azotées absorbent toutes dans l’UV à 260 nm environ.
Absorption de la molécule d’ADN est nettement inférieure à celle que l’on obtiendrait avec un mélange des mêmes bases libres aux mêmes concentrations
car le coefficient d’extinction molaire ε des bases libres est bien supérieur à celui des bases appariées (si ε diminue pour une même concentration (c), A diminue aussi)
Donc cette différence d’absorption (environ 30%) entre ADN et mêmes bases libres est due à
l’appariement des bases par liaison H.
Ce phénomène est appelé effet hypochrome.
Propriétés spectrales
Autre phénomène: hyperchromie Les solutions d’ADN présentent
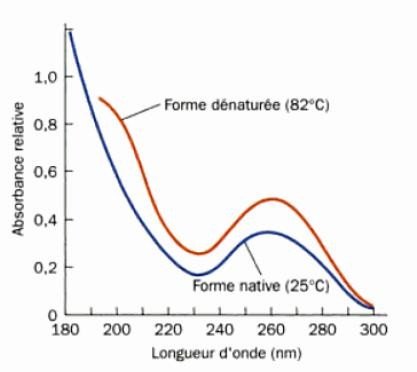
le maximum d’absorption à la
longueur d’onde de 260 nm.
MAIS pour une longueur d’onde donnée, l’ADN monocaténaire absorbe plus que l’ADN bicaténaire.
Dans l’ADN double brin, les bases sont masquées ou se chevauchent, alors que dans l’ADN simple brin il n’y a pas de structure qui cache les bases, donc l’absorbance est plus importante.
Si on chauffe une solution d’ADN bicaténaire à différentes températures et qu’on suit l’absorbance de cette solution pour chacune de ces T° : on peut construire une courbe A=f (T°C de traitement) = courbe de dénaturation thermique de l’ADN.
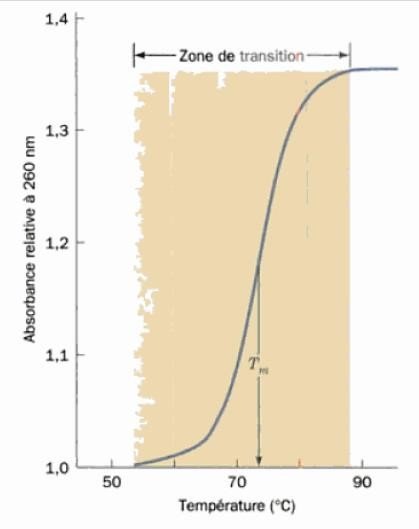
Observation : l’absorbance de l’ADN augmente avec la température.
L’ADN monocaténaire présente une absorption plus importante qu’un ADN bicaténaire.
La courbe a une allure sigmoïde.
Le point d’inflexion de cette courbe correspond, sur l’axe des abscisses, au Tm (Melting temperature).
Tm = température de fusion de l’ADN
= température moyenne pour laquelle la moitié de cet ADN est dénaturé.
Dénaturation diminution de la viscosité de la solution
augmentation de la densité de l’ADN.
La dénaturation de l’ADN :
–augmentation de l’absorption dans l’ultra-violet
–diminution de la viscosité
– et augmentation de
la densité.
ACIDES NUCLEIQUES
Dénaturation de l’ADN

Densité optique 260 nm
| ADNmonocaténaire | |
| ADN bicaténaire |
Tm Température (°C)
260nm
ADN double brin
260 nm
ADN simple brin
<
67
ADN: Absorbance
68
Les acides nucléiques absorbent à ~260 nm (à cause des bases puriques/pyrimidiques);
Les préparations d’acides nucléiques pures donnent un
rapport A260/A280 d’environ1.8;
Des valeurs de A260/A280 inférieures à 1.8 sont généralement indicatives de la contamination des acides nucléiques par des protéines.
une absorbance de 1 à 260 nm donne:
50 µg / ml d’ADN
40 µg / ml d’ARN

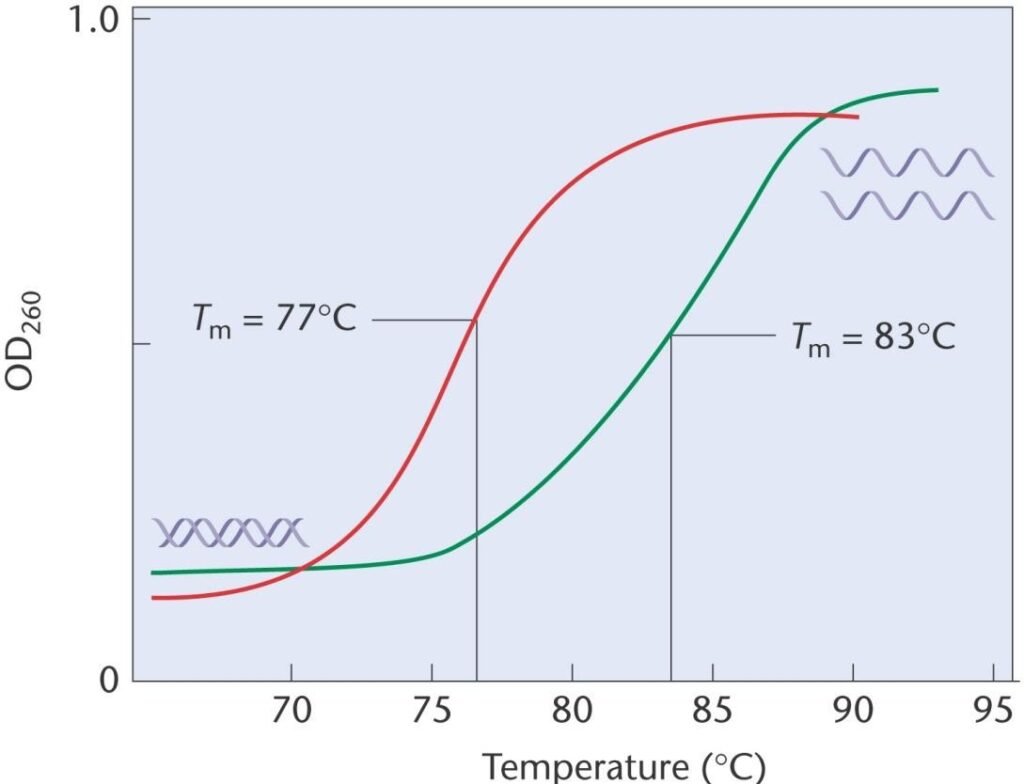
La température à laquelle 50% de l’acide nucléique db s’est dénaturé est appelée la température de fusion (Tm)
Dénaturation de l’ADN
DNA#1
DNA#2
Le Tm est affecté par plusieurs
facteurs:
Concentration en sels.
Longueur des molécules: Tm augmente
Hyperchromicité
avec la longueur (pour ADN < 150 pb)
Contenu en G+C: Plus le contenu en GC est élevé, plus le Tm sera aussi élevé.
69
Dénaturation/Renaturation
Dénaturation d’une molécule = perte de
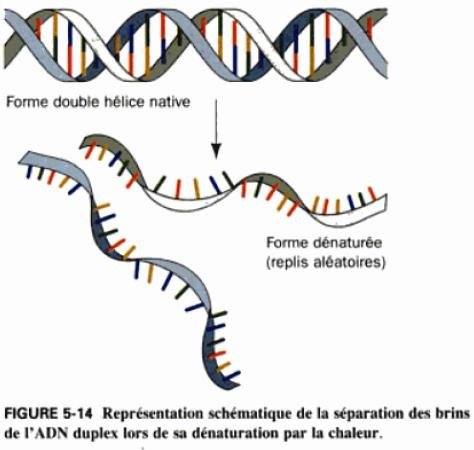
sa structure tridimensionnelle sans altération
de la structure primaire.
Pour une molécule d’ADN double brins
= rupture des liaisons hydrogène entre les
bases : on obtient de l’ADN monocaténaire.
Phénomène coopératif
sel)
On peut obtenir une dénaturation de l’ADN par
des moyens physiques (température, pH extrême, diminution de la concentration en
et des moyens chimiques (utilisation de l’urée, de soude, de formaldéhyde).
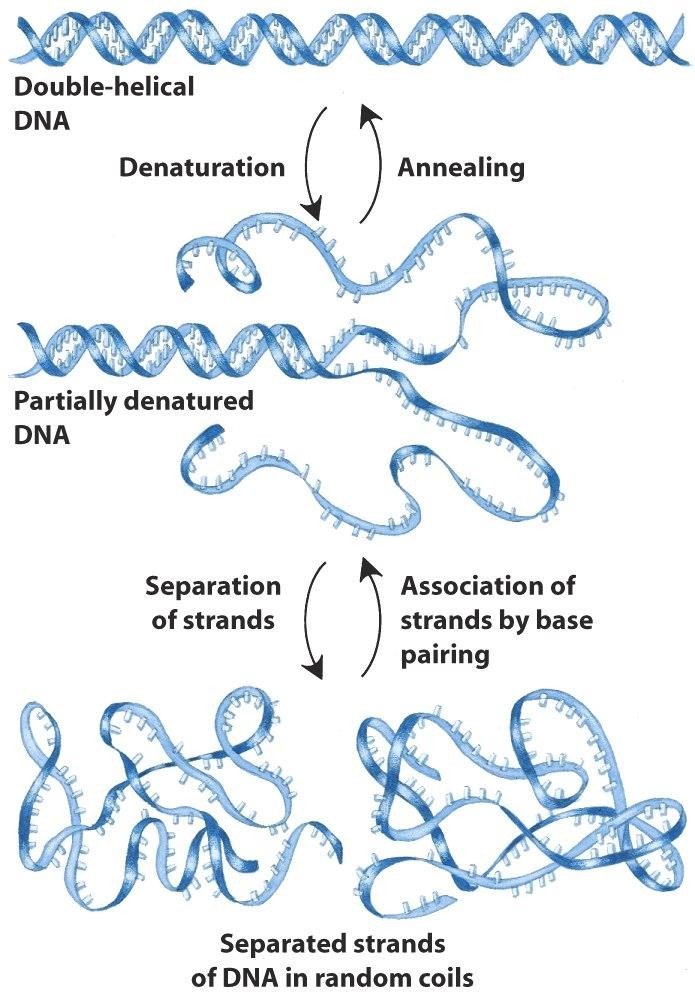
Les acides nucléiques double brins (db) (ds) peuvent être convertis en acides nucléiques simple brins (sb) ( dénaturés) de plusieurs façons:
Dénaturation de l’ADN
Augmentation de la température
Diminution de la concentration de sel
Produits chimiques: NaOH/formamide/formaldéhyde (brisent les ponts H)
Inversement, l’ADN sb ou SS peut être
renaturé de la façon suivante: :
Diminution de la température
Augmentation de la concentration en sel
Ce phénomène Denaturation-Renaturation
peut être suivi par spectrophotométrie:
Les acides nucléiques sb absorbent davantage à 260 nm que les acides nucléiques db: hyperchromicité;
Renaturation = Hypochromicitté
71
Température et Renaturation de l’ADN
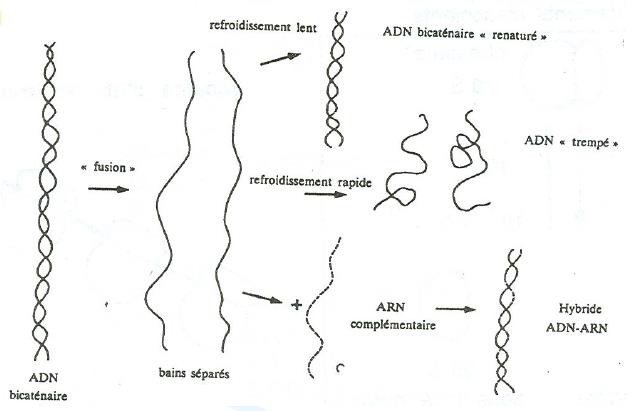
Si une solution d’ADN dénaturé est refroidie rapidement bien en dessous de sa Tm, l’ADN résultant ne sera que très partiellement apparié car les brins complémentaire n’ont pas le temps de se réassocier convenablement.
Cependant si on refroidit lentement la solution d’ADN dénaturé, l’ADN se renature complètement.
De la même façon, des brins complémentaires d’ADN et d’ARN peuvent s’hybrider pour former une double hélice.
Hybridation des acides nucléiques et des sondes nucléiques
L’hybridation est une propriété fondamentale des acides nucléiques qui repose sur les règles de complémentarité.
Il est possible d’apparier des brins d’ADN (ou d’ARN) avec des oligonucléotides ou polynucléotides qui reconnaissent
spécifiquement des séquences sur les brins d’ADN de manière anti-parallèle et complémentaire.
Ces oligo ou polynucléotides sont appelés sondes nucléiques
Remarque : Oligonucléotide (oligomère) < 50 nucléotides
73
Hybridation des AN
74
Hybridation des AN
Les acides nucléiques sb ayant des séquences complémentaires vont se
renaturer lorsqu’elles seront mélangées ensemble (hybridation);
ADN-ADN
ADN-ARN
ARN-ARN
La renaturation se produira même si les deux brins ne sont pas parfaitement complémentaires
Cependant, le Tm diminue avec le nombre de différences dans la complémentarité (mismach)
Ce phénomène est très utilisé lors de l’étude des acides nucléiques (techniques de BM) :
Séquençage
PCR
Analyse Southern
Analyse Northern
Analyse FISH
Puces à ADN
75
Paramètres influençant la dénaturation
Extraction, Purification et quantification de l’ADN
Dans un premier temps, on doit détruire la structure cellulaire du tissu par broyage puis on isole les noyaux par centrifugation.
Dans un deuxième temps, on extrait et on purifie l’ADN des noyaux par élimination successives des protéines associées, des lipides et de l’ARN.
Il existe de nombreuses techniques de purification des acides nucléiques comme :
La chromatographie sur colonne de gel en présence d’un dénaturant
La chromatographie d’adsorption
Le plus souvent on se contente de multiples précipitations par l’éthanol qui suffisent à débarrasser l’ADN des protéines contaminantes et des enzymes.
Effets des acides et alcalis sur les A.N
Acide fort = augmentation de la T°= hydrolyse complète de AN.
Acide faible = hydrolyse ménagée = coupure de la liaison N-osidique : AN apuriniques ou apyrimidiniques;
Hydrolyse alcaline: changement tautomérique:
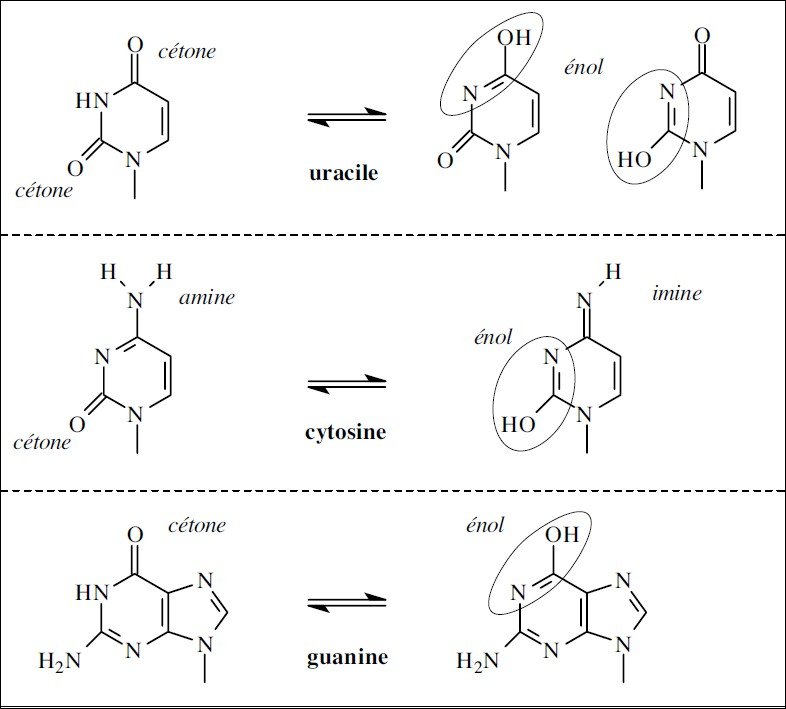
🞑 PH neutre : forme ceto (oxo)( forme physiologique.
🞑 PH alcalin : forme enol
HYDROLYSE ENZYMATIQUE des Acides Nucléiques
79
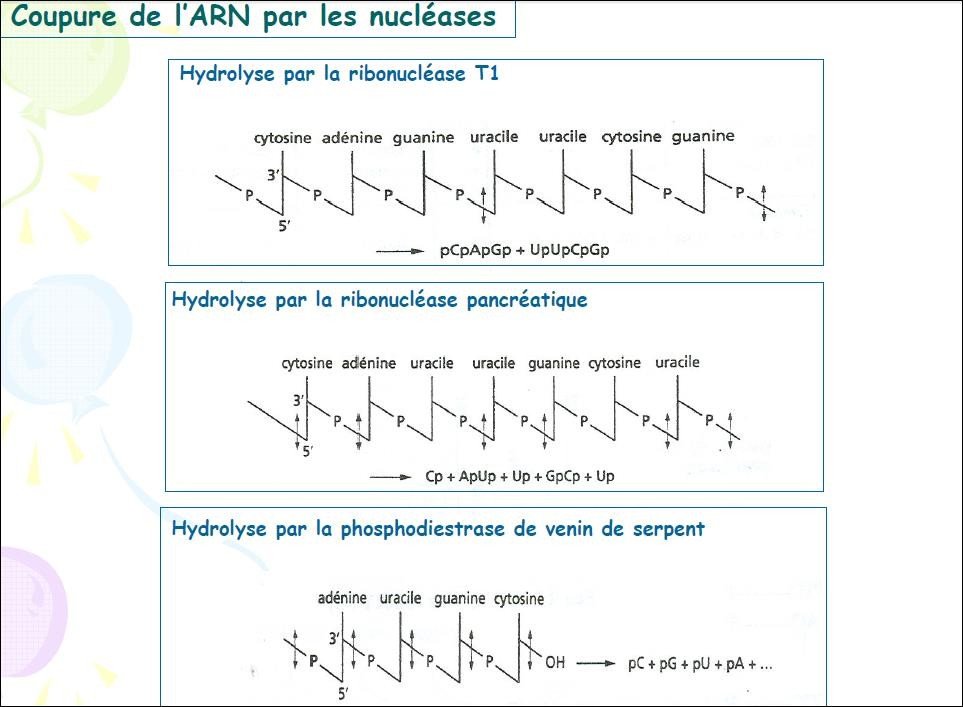

Coupe en b après une guanine
Coupe en b après une PY
Coupe en a
80
ENZYMES de RESTRICTION
Les enzymes de restriction sont des hydrolases agissant sur des liaisons esters : sont des estérases.
Elles sont produites par les bactéries lorsqu’elles sont infectées par un bactériophage.
Ce Sont des ciseaux moléculaires qui hydrolysent l’ADN double brin
81
ENZYMES de RESTRICTION
Les enzymes de restriction sont des ciseaux moléculaires qui hydrolysent l’ADN,
Il existe 3 types d’enzymes classés en fonction des sites qu’elles reconnaissent:
enzymes de type I :
l’enzyme reconnaît un site particulier et coupe à environ 1000 jusqu’à 5000 nucléotides plus loin.
enzymes de type II :
ce sont les plus nombreuses et les plus utilisées aux laboratoires de BM.
Leurs sites de restrictions de 4 à 8 paires de bases sont des séquences palindromiques.
enzymes de type III :
L’enzyme reconnaît une séquence mais coupe à une vingtaine de paires de bases plus loin.
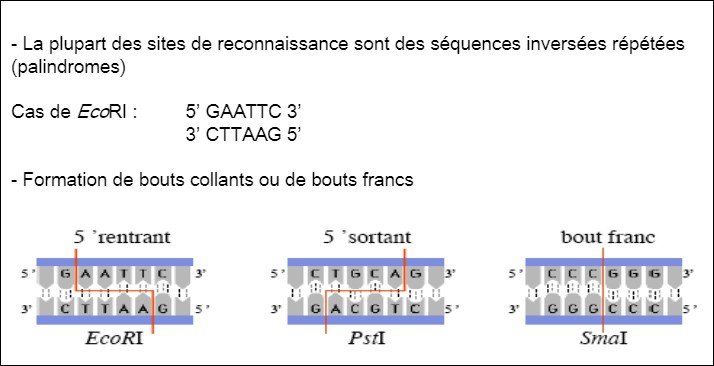
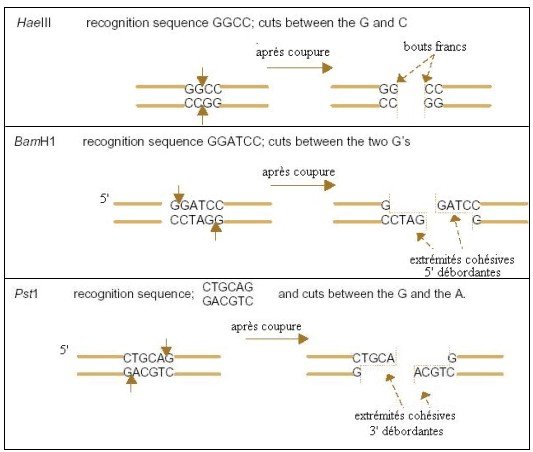
Liste non exhaustive des enzymes de restriction
85
86
Applications des enzymes de restriction : clonage moléculaire
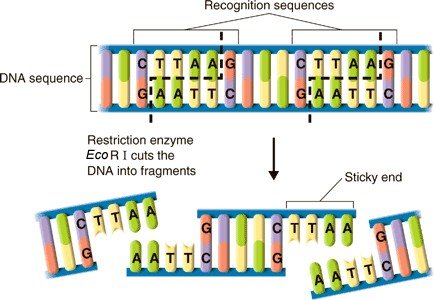
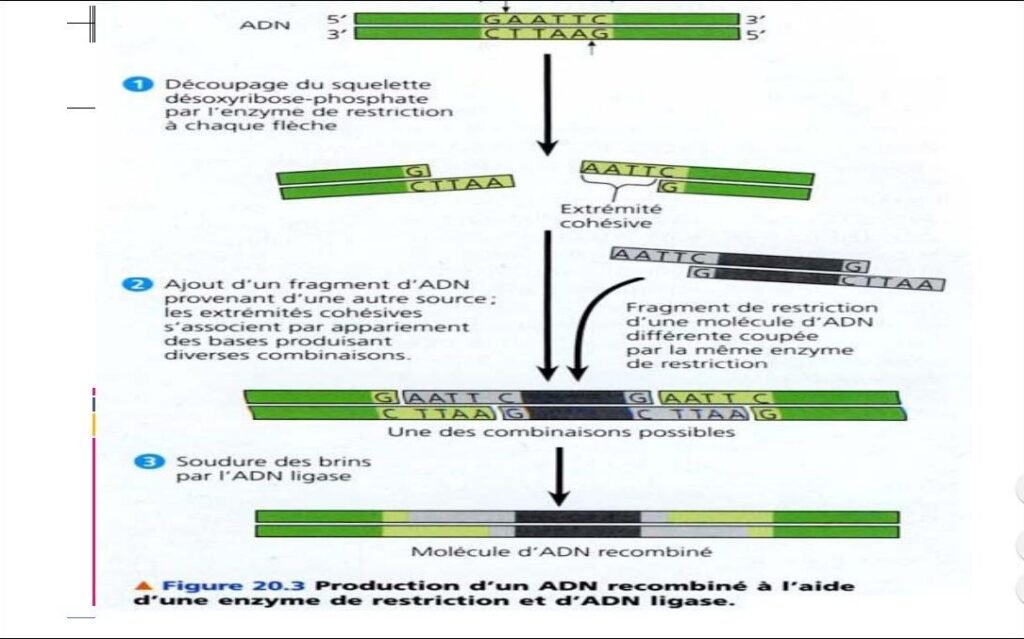
87
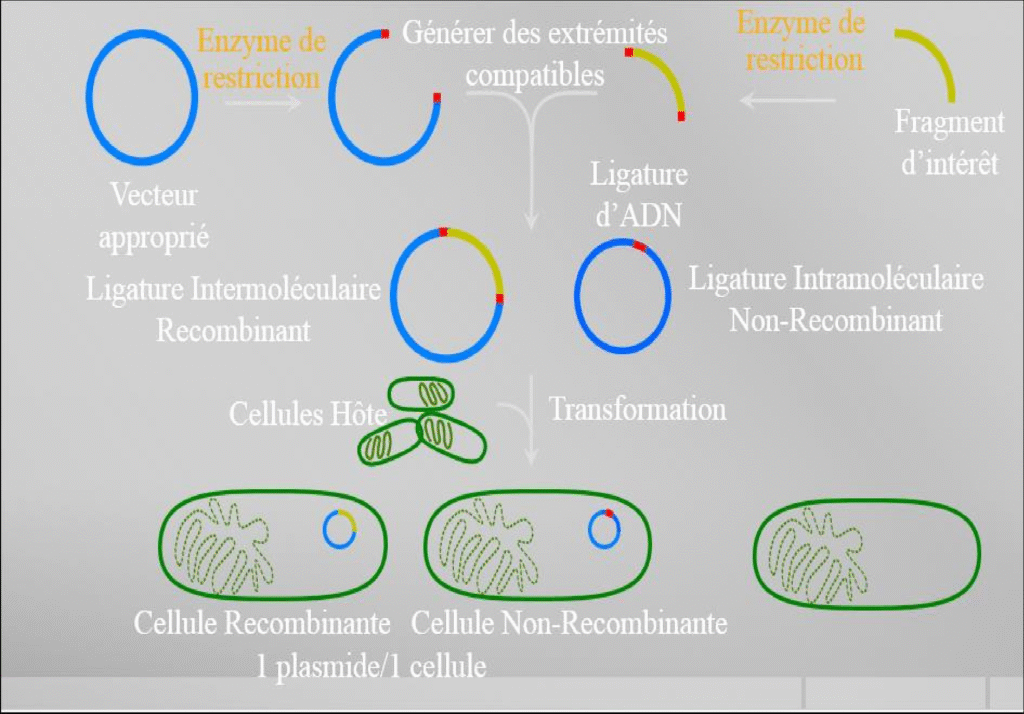
88
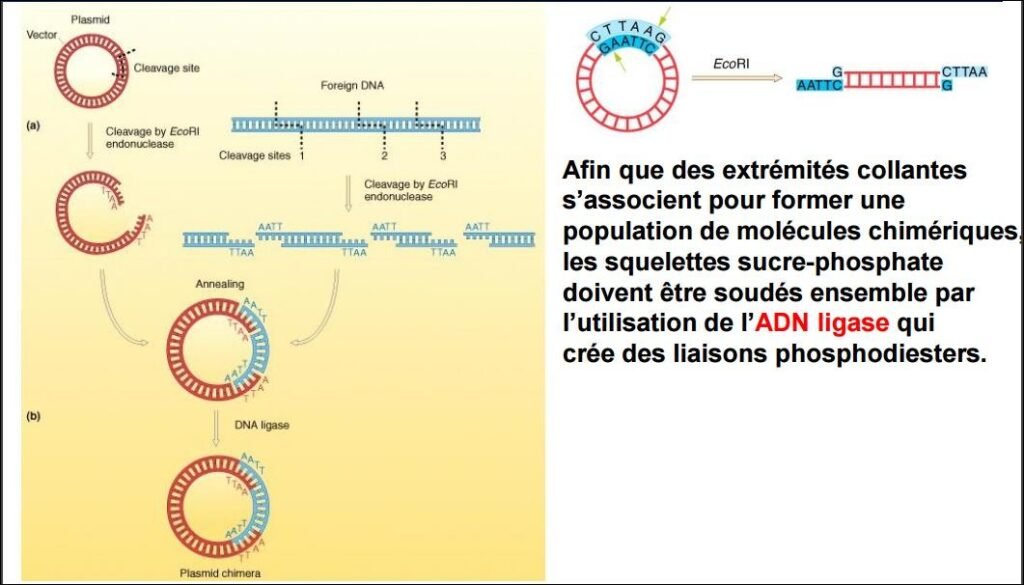
89
Applications : clonage moléculaire
Domaines applications
Protéines recombinantes (médicaments):
Insuline
Hormone de croissance
Albumine
Facteur VIII
Erythropoïétine (EPO)
Interféron
Animaux transgéniques (modèles Mdies)
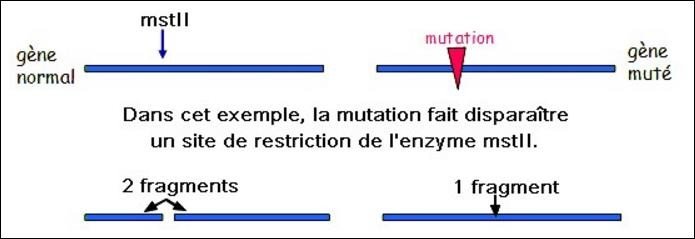
On distingue des sites de restriction obligatoires (toujours présents) et des sites de restriction variables
Mst II
1 2 3 4
Polymorphisme
5
20Kb
Après digestion avec la même enzyme, la longueur des fragments diffère d’un individu un autre
🞹
Variablité par la taille des fragments qui constitue un polymorphisme de restriction = RFLP (Restriction Fragment Lengh Polymorphism) : peut être utilisé comme marqueur génétique
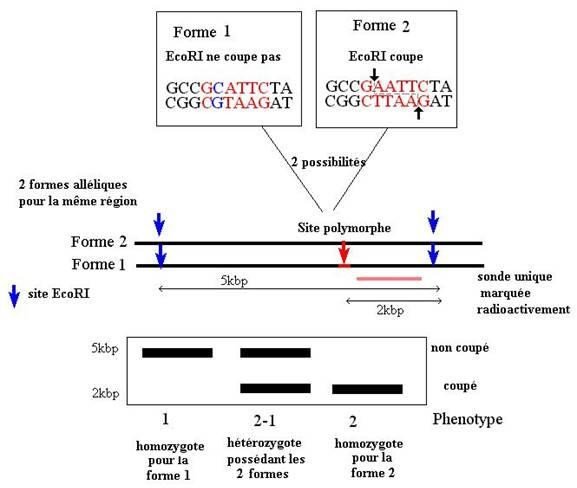
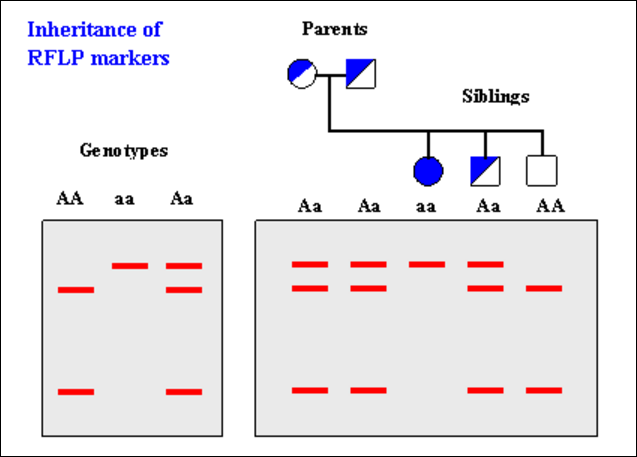
92
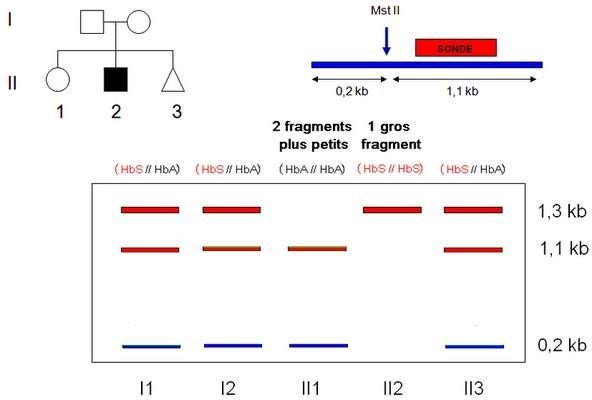
Application des enzymes de restrictions : Diagnostic moléculaires des maladies génétiques
Séparation des fragments par électrophorèse (en fonction de leurs tailles)
Comment les enzymes de restriction peuvent-elles contribuer au dépistage d’un allèle muté : Ex de la drépanocytose, maladie génétique liée à la formation d’une hémoglobine anormale HbS
93

